Revista Digital Universitaria ISSN: 1607 - 6079 | Publicación mensual
1 de diciembre de 2014 vol.15, No.12
• TEMA DEL MES •
Enzimas: aceleradores de reacciones químicas en las células y en la industria
Introducción
La definición del diccionario de biocatalizador es: “aquella sustancia, en especial una enzima, que inicia o modifica la velocidad de una reacción química sin ser ella misma afectada por esta reacción”. Efectivamente, las enzimas son macromoléculas (normalmente proteicas) que tienen la capacidad de aumentar la velocidad de las reacciones en millones de veces comparadas con el tiempo que les tomaría ocurrir espontáneamente.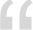 | Las enzimas o biocatalizadores son energéticamente eficientes y prácticamente todas llevan a cabo su función bajo condiciones moderadas: temperatura ambiente, presión atmosférica y en ambientes acuosos. | |
 | ||
Al emplear técnicas que intentan imitar la evolución natural en un proceso de generación de variabilidad-selección, la ingeniería de proteínas utiliza diferentes estrategias dependiendo del grado de conocimiento que se tenga sobre ellas. De una enzima es posible conocer la secuencia del gen que la codifica, que es indicativa de su secuencia de aminoácidos, según el código genético. A esta secuencia lineal de aminoácidos se le denomina estructura primaria. La posición que los diferentes aminoácidos adoptan puede resultar en la formación de hélices o láminas conocidas como alfa-hélices u hojas-beta plegadas. En su conjunto, a éstas se les conoce como estructura secundaria. La forma tridimensional del acomodo completo de todos los aminoácidos en el espacio se conoce como estructura terciaria (Figura 1), y se determina experimentalmente, entre otras maneras, mediante difracción de rayos X de arreglos cristalinos de las proteínas. Por esta razón es que comúnmente se puede llamar también a la estructura tridimensional de una proteína, estructura cristalina1.
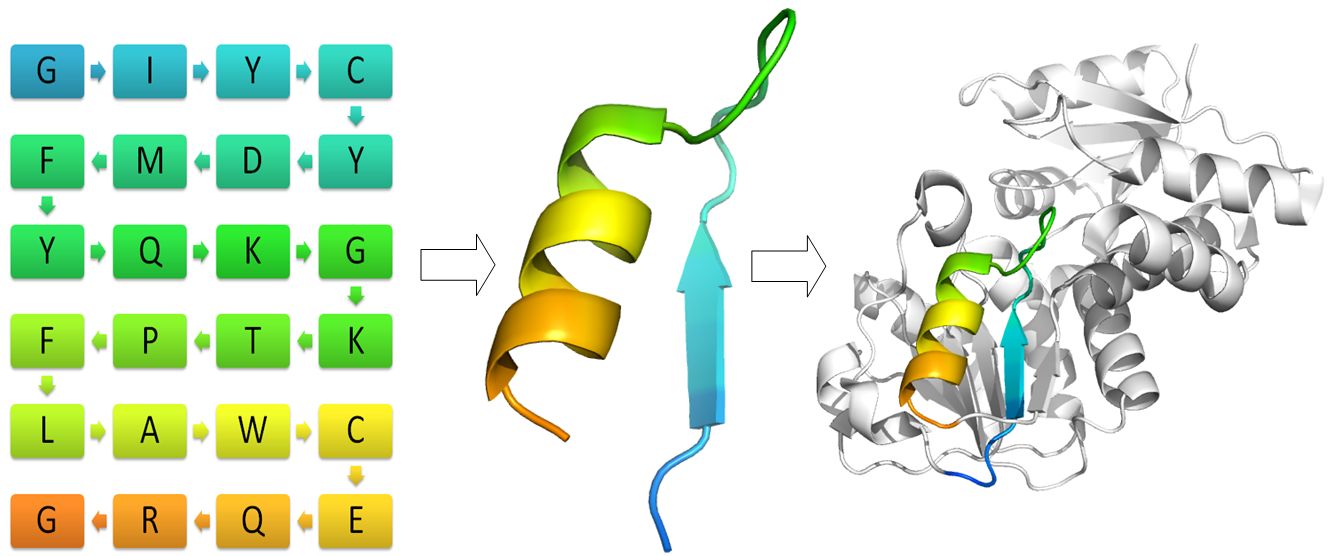
Figura 1. Se muestra la secuencia lineal (indicada por las flechas pequeñas) que corresponde a la estructura primaria de una proteína. Cada bloque indica el nombre del aminoácido con el código de una sola letra, ejemplo: G = glicina. El color de esta secuencia de aminoácidos permite visualizar su participación en la formación de hélices u hojas (estructura secundaria), así como el lugar que éstos ocupan en el espacio tridimensional dentro de toda la proteína..
Aunque la determinación de la estructura tridimensional de las proteínas se ha vuelto más cotidiana, solamente un pequeño porcentaje de las enzimas con las que se trabaja en los laboratorios cuenta con datos estructurales. Por este motivo, es muy común tener datos de la secuencia primaria y secundaria de una proteína, pero no siempre se conoce la estructura terciaria. Esta información determina el método que se quiera aplicar para llevar a cabo el mejoramiento de una enzima. En general, los métodos de ingeniería de proteínas pueden dividirse ampliamente en dos tipos: a) métodos de evolución dirigida, y b) métodos de diseño racional. En muchos casos exitosos la combinación de ambos enfoques ha permitido mejores resultados.
1 Se puede encontrar más información al respecto de la estructura de las proteínas y de los métodos para su análisis en: http://laguna.fmedic.unam.mx/~evazquez/0403/proteinas.html.
* Un ejemplo ilustrativo de la evolución dirigida de proteínas se puede ver en el siguiente video animado: https://www.youtube.com/watch?v=r7mSweNf54c
Evolución dirigida
Cuando la información estructural de una enzima es limitada, la producción adecuada de la proteína en el laboratorio es suficiente para llevar a cabo ensayos de evolución dirigida. En este caso, la teoría de la evolución conocida para las especies, en la que la acumulación de mutaciones genera variabilidad, es aplicada en la evolución dirigida de proteínas, aunque en escalas de tiempo miles de veces más cortas.Para este propósito el gen de la enzima de interés es sometido a la técnica conocida como PCR (por sus iniciales en inglés: polymerase chain reaction), en la que el fragmento de DNA que codifica a la enzima es copiado y replicado millones de veces por una enzima polimerasa de DNA. Si esta polimerasa replica al DNA introduciendo errores que no son reparados, al final de la reacción de PCR se tendrán millones de moléculas portadoras de cambios (o mutaciones) con respecto a la original. Cuando se desconoce qué secuencia de aminoácidos es la que conferirá una mejor actividad a la enzima, es deseable que los cambios que se producen en la reacción de PCR ocurran al azar y con poca frecuencia (para evitar generar muchas variantes inactivas) (Figura 2).
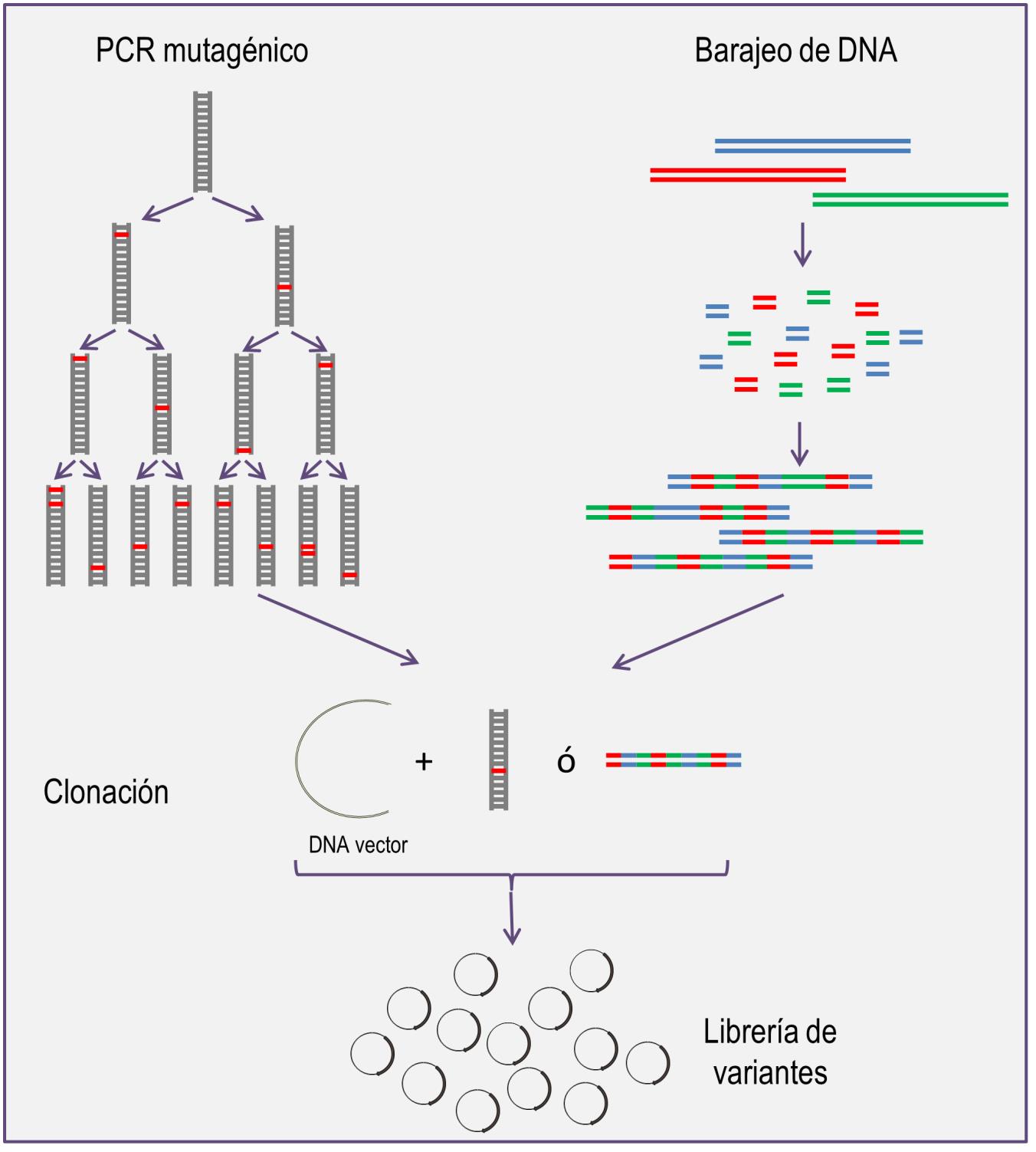
Figura 2. Mediante PCR mutagénico se genera variabilidad a partir de la secuencia de un solo gen. Con esta técnica la molécula parental (indicada en gris) es replicada a través de muchos ciclos (aproximadamente 30), y cada vez son introducidos errores en diferentes posiciones (indicados con líneas rojas). El método de barajeo de DNA involucra el corte de varias secuencias similares (indicados por líneas dobles de diferente color) y su posterior re-ensamblaje al azar originando genes compuestos de diferentes fragmentos parentales (mostrados en líneas dobles de colores combinados). Después, las moléculas obtenidas por cualquiera de estas dos formas son clonadas en un fragmento de DNA llamado vector en el que se liga cada fragmento modificado para generar las librerías conteniendo miles de variantes.
Las técnicas actuales en el laboratorio permiten manipular o controlar la tasa de error durante la replicación de un gen, pudiendo llegar a tener de 2 a 8 mutaciones por cada 1000 pares de bases replicadas. Las poblaciones de moléculas variantes que llegan a obtenerse con este método deben ser posteriormente clonadas en un vector para su producción y la verificación del cambio enzimático buscado (Figura 3). Los genes de la o las variantes que presenten cambios positivos pueden ser sometidos a nuevos ciclos de PCR para intentar obtener aún mejores actividades. Cuando la mutagénesis ocurre a lo largo de toda la secuencia proteica, el número de variantes generadas es inmenso y en la práctica es posible el análisis de sólo una fracción infinitesimal de estas colecciones (también llamadas librerías).
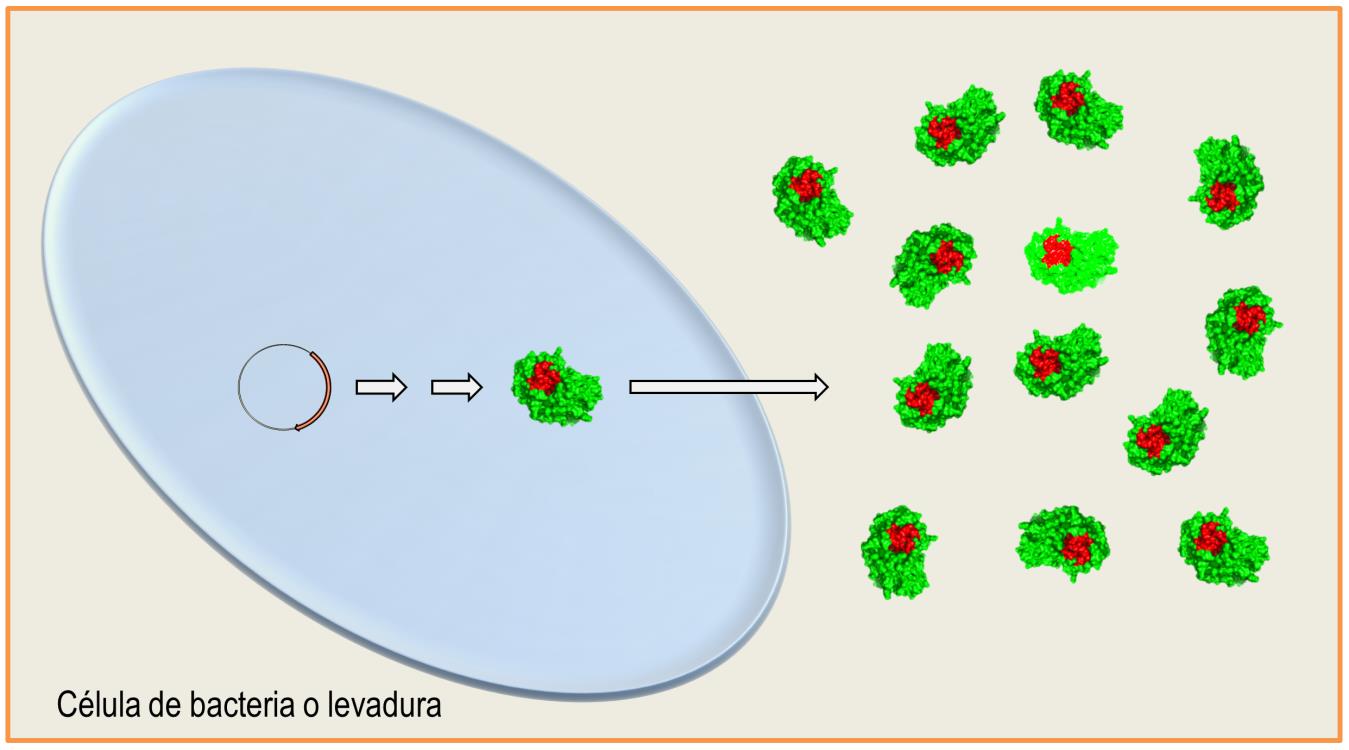
Figura 3. Las librerías de mutantes son introducidas dentro de células –que pueden ser de bacterias o levaduras, por ejemplo– para que, mediante los mecanismos celulares (indicados por las flechas pequeñas), sean producidas enzimas con pequeños cambios respecto a la proteína original (en este caso se ilustra una proteína de color pálido como aquella que por azar resultó con una mejor actividad). La flecha larga indica la producción en alta cantidad de la enzima y su exportación hacia afuera de la célula.
Otra manera en la que puede llevarse a cabo la evolución dirigida es mediante barajeo de DNA que genera diversidad mediante la combinación de secuencias de genes similares que existen naturalmente. Las colecciones de DNA recombinantes son generadas mediante la fragmentación al azar de los genes homólogos, seguida del ensamblaje de los fragmentos, lo que genera entrecruzamientos en las áreas en las que existe identidad en la secuencia.
El éxito de las técnicas de evolución dirigida depende de contar con un método que permita la identificación de la variante con la actividad deseada de entre los millones de mutantes generados en una sola librería. Debido a que no todas las enzimas son sujeto para el desarrollo de sistemas de búsqueda sencilla dentro las librerías, en muchas ocasiones el método para la selección de las variantes mejoradas es difícil de implementar, y esto constituye la principal desventaja de la evolución dirigida al azar. Por esta razón, los esfuerzos actuales se enfocan en la obtención de librerías inteligentes que produzcan un número de variantes mucho menor mediante la identificación de los mejores aminoácidos candidatos a la mutagénesis. Por ejemplo, las zonas de funcionalidad de una proteína pueden ser identificadas usando información evolutiva que permite la identificación de aquellos aminoácidos que son comunes entre genes de diferentes organismos, y que indican que su posición en la secuencia o en la estructura es importante, lo que delimita la mutagénesis a estas zonas blanco haciendo más sencilla la búsqueda de las variantes mejoradas.
Diseño racional
El diseño racional se refiere a la capacidad de predecir los cambios requeridos para lograr alguna función preconcebida de una enzima. Esta estrategia requiere de un conocimiento profundo de la estructura de una enzima y de su sitio activo, que es el lugar en donde se lleva a cabo la catálisis (Figura 4), o de los cambios moleculares que ocurren durante la reacción, debido a que la función está estrechamente relacionada a la estructura.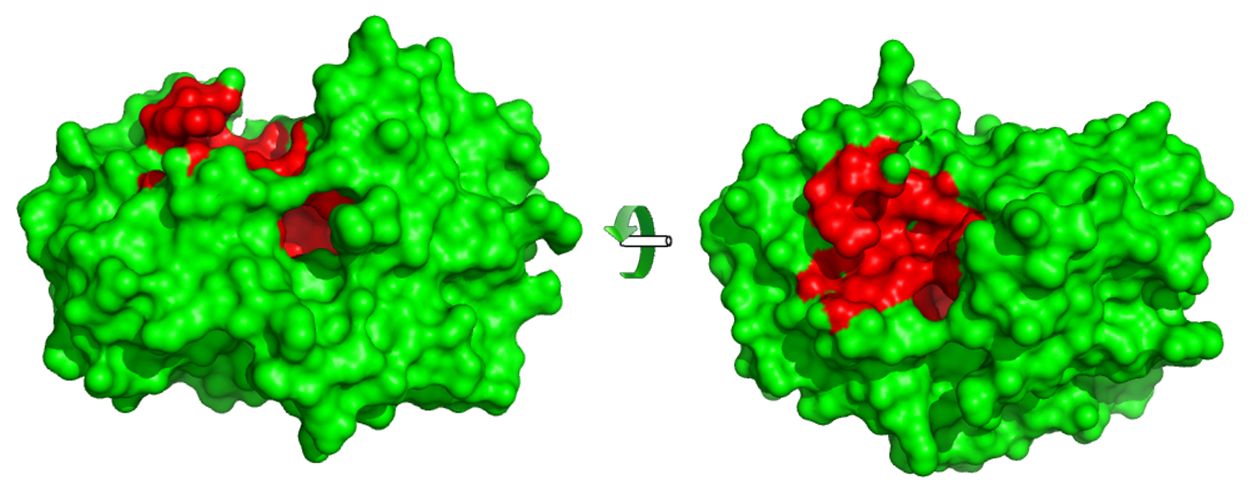
Figura 4. Estructura tridimensional de una proteína, en la que se ha marcado con color rojo el sitio activo. A la izquierda se muestra una vista lateral y a la derecha una vista de frente de la enzima.
Como se mencionó anteriormente, la determinación de las estructuras cristalográficas de muchas proteínas ha permitido que las bases de datos en donde se deposita esta información estén en constante crecimiento. Por otro lado, existen herramientas computacionales cada vez más poderosas que permiten la generación de modelos tridimensionales de proteínas para las cuales no hay datos cristalográficos, y que se basan en
 | ...existen herramientas computacionales cada vez más poderosas que permiten la generación de modelos tridimensionales de proteínas para las cuales no hay datos cristalográficos, y que se basan en la información de proteínas similares cuya estructura ya ha sido determinada. | |
 | ||
Video 1. Visualización de enzimas.
Casos de éxito de proteínas mejoradas
El número reportado de enzimas mejoradas mediante ingeniería de proteínas es grande, y existen ejemplos para diferentes propósitos tales como: enzimas con usos terapéuticos, industriales o medioambientales. A continuación se describen tres ejemplos del mejoramiento de enzimas utilizando diferentes métodos.Ejemplo 1
Para eliminar restos biológicos de la ropa, algunos detergentes incorporan en su fórmula enzimas como proteasas, lipasas o amilasas. Las enzimas añadidas deben ser estables en las condiciones de lavado que incluyen la presencia de surfactantes, agentes oxidantes y, en ocasiones, altas temperaturas. | La subtilisina es una proteína con actividad de proteasa –es decir, que destruye otras proteínas– que probó ser útil al ser incorporada en detergentes... | |
 | ||
Ejemplo 2
En este estudio se requería el cambio de especificidad de sustrato de una enzima, es decir, que ésta prefiriera actuar sobre un compuesto diferente al que transforma naturalmente. El objetivo fue invertir la especificidad de la enzima xilosa reductasa de una levadura llamada Pichia stipitis, que naturalmente utiliza NADP como cofactor, para que en su lugar utilizara NAD en la reacción. Este cambio disminuiría el desbalance oxidativo poco favorable en la producción de alcohol a partir de xilosa. Este trabajo tuvo un enfoque semi-racional usando una combinación de diseño racional y evolución dirigida. La estrategia utilizada consistió en el análisis de la estructura tridimensional de la proteína para identificar y hacer una lista de los aminoácidos que están involucrados en el reconocimiento del cofactor(KRAHULEC et al., 2009).Al momento del estudio, la enzima xilosa reductasa no contaba con datos cristalográficos de estructura, pero fue posible predecir un modelo estructural por su parecido a proteínas homólogas con estructura conocida. En el modelo estructural se identificaron 6 aminoácidos involucrados en el reconocimiento del cofactor lo que los convertía en candidatos a ser modificados. Dos de los sitios candidatos se mutaron al azar simultáneamente en una primera ronda de mutación, y la variante con mejor actividad se eligió como molde de una segunda ronda de mutación donde se cambiaron otros dos sitios de la lista inicial. Una tercera ronda de mutaciones se realizó con la mejor mutante de la segunda ronda. La variante final pasó de preferir dos veces NADP a preferir 25 veces NAD. Esta estrategia se basó en el uso de la información estructural de la proteína lo que redujo considerablemente el número de variantes posibles, lo cual fue de gran ayuda debido a que el método de selección no permitía abarcar el análisis de muchas variantes.
Ejemplo 3
En este ejemplo se abordó el problema de la degradación de compuestos tóxicos introducidos al ambiente por procesos industriales y agrícolas mediante la estrategia de barajeo de DNA. Como sistema, se eligieron dos enzimas llamadas atzA y triA, que presentan solamente nueve aminoácidos de diferencia entre sí. Éstas inician la hidrólisis de los compuestos s-triazinas, una mediante la eliminación de cloro (desclorinación) y la otra mediante la eliminación del grupo amino (desaminación) de los compuestos atrazina y melamina, respectivamente. | Las s-triazinas comenzaron a usarse recientemente como herbicidas, pesticidas o en plásticos, por lo cual su liberación en el ambiente ha sido extensa. | |
 | ||
Conclusión
El uso de biocatalizadores podría satisfacer la creciente demanda de procesos industriales limpios. Sin embargo, la producción de enzimas a gran escala es todavía costosa. El uso de enzimas que resistan, por más tiempo, las diferentes condiciones operacionales, podría ayudar a aliviar este problema. La ingeniería de proteínas es una alternativa para la producción de actividades específicas tomando como base lo que la naturaleza ha creado. El conocimiento cada vez mayor sobre la genética y bioquímica de las proteínas, junto con el número creciente de secuencias de proyectos de secuenciación masiva y que se encuentran públicamente disponibles, y sobre la estructura de diferentes tipos de proteínas, han facultado el desarrollo de técnicas que en su conjunto permiten identificar residuos clave para la manipulación y confección de enzimas con características mejoradas.Glosario de términos
Aminoácido: Bloque de construcción de las proteínas que se une con otro en sus extremos para formar cadenas. Existen 20 aminoácidos diferentes en las proteínas encontradas en la naturaleza.Proteína: Cadena compuesta por los 20 aminoácidos con un orden específico indicado por el gen que la codifica.
Secuencia: Es el orden en que se encuentran los aminoácidos (que forman las proteínas) o los nucleótidos (que forman el DNA) en una cadena lineal.
Residuo o residuo de aminoácido: Aminoácido que forma parte de la secuencia de una proteína.
DNA: También llamado ADN, es la molécula que guarda la información para especificar la secuencia de las proteínas (entre otras funciones) en los seres vivos.
Gen: Cadena de DNA que contiene la información que indica la secuencia específica de una proteína.
Polimerasa de DNA: Un tipo particular de enzima cuya función es replicar nuevas cadenas de DNA.
Mutación: Cambio en la secuencia de la cadena de DNA.
Mutagénesis: Generación de mutaciones en el DNA.
Homólogo: Proteína que tiene cierto parecido en su secuencia con otra.
Catálisis: Aceleración de una reacción química.
Lipasas, proteasas, amilasas: Proteínas que degradan grasas, proteínas y almidón, respectivamente.
Surfactantes: Agentes químicos que humedecen, emulsifican y dispersan compuestos cuando se disuelven en agua.
Agentes oxidantes: Son compuestos que aceptan electrones en una reacción de oxidación/reducción.
Serina, alanina, metionina: Nombres de tres aminoácidos de los 20 encontrados en las proteínas.
NADP, NAD: Fosfato de nicotinamida adenina dinucleótido y nicotinamida adenina dinucleótido, respectivamente. Son sustancias que actúan en conjunto con algunas proteínas para permitirles llevar a cabo reacciones de oxidación/reducción.
Cofactor: Moléculas que actúan en conjunto con algunas proteínas para permitirles llevar a cabo su función, como el NADP o el NAD.
Reductasa: Enzima que acelera reacciones de oxidación/reducción.
Hidrólisis: Reacción química que consiste en la ruptura de un enlace con la formación de una molécula de agua en el proceso.
Bibliografía
PEÑA-MONTES, C. y A. Farrés Gonzalez-Saravia, "Evolución Dirigida en la generación de biocatalizadores: Biocatalizadores hechos a medida". BioTecnología, 2008. 12(2): p. 5-23. SEGOVIA, L. y M. Peimbert, "Ingeniería de Proteínas y Evolución Dirigida". Mensaje Bioquímico, 2010. XXXIV: p. 135-141. ESTELL, D.A., T.P. Graycar, y J.A. Wells, "Engineering an enzyme by site-directed mutagenesis to be resistant to chemical oxidation". Journal of Biological Chemistry, 1985. 260(11): p. 6518-6521. KRAHULEC, S., M. Klimacek, and B. Nidetzky, "Engineering of a matched pair of xylose reductase and xylitol dehydrogenase for xylose fermentation by Saccharomyces cerevisiae". Biotechnology Journal, 2009. 4(5): p. 684-694.RAILLARD, S., et al., "Novel enzyme activities and functional plasticity revealed by recombining highly homologous enzymes". Chemistry & Biology, 2001. 8(9): p. 891-898.
COMENTARIOS
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO
Dirección General de Cómputo y de Tecnologías de Información y Comunicación
Dirección General de Cómputo y de Tecnologías de Información y Comunicación
2013 Esta obra está bajo una licencia de Creative Commons