Revista Digital Universitaria ISSN: 1607 - 6079 | Publicación mensual
1 de enero de 2015 vol.16, No.1
• TEMA DEL MES •
Estructura, función y organización de las proteínas. Un Homenaje al Dr. Armando Gómez Puyou
Introducción
En una tarde de mediados del año 2002, recibí una llamada telefónica del Dr. Armando Gómez Puyou. Me dijo que recordaba haber escuchado una conferencia que yo había dado un año antes acerca del efecto hidrofóbico, y me preguntó si podríamos reunirnos a discutir algunos resultados de uno de sus alumnos de doctorado que, él pensaba, podrían explicarse empleando ese concepto. Nos encontramos en su oficina, donde la discusión de los datos de su alumno –de la inhibición de una proteína debido a su unión a una molécula pequeña– derivó rápidamente en una plática general sobre las interacciones entre proteínas y ligandos, un tema en el que yo estaba haciendo mis pininos en mis esfuerzos por moverme hacia esa área de investigación. La reunión, en la que aprendí mucho más del tema que de todos los artículos que había leído los meses anteriores, terminó cuando le dije que podíamos intentar comprender mejor los datos de su alumno empleando un equipo que yo tenía en mi laboratorio, un calorímetro, y él respondió con un "vamos a hacerlo". Ahí inició una colaboración y una amistad que sólo terminó con su muerte hace unos meses. En este espacio, relataré un par de esas colaboraciones.¿Qué es y cómo mide un calorímetro?
Un calorímetro es un instrumento que sirve para medir la cantidad de calor que libera o absorbe una muestra en la que ocurre algún evento que refleja las interacciones moleculares presentes entre los componentes de la muestra. Éste puede ser la unión de una molécula pequeña (ligando) a una macromolécula (proteína), una reacción química o el cambio estructural que sufre una proteína cuando se calienta. Todos los calorímetros constan de los mismos elementos básicos en su diseño y operan con los mismos principios. Las diferencias entre ellos están en la cantidad de muestra que requieren y en su precisión.La Figura 1 muestra los elementos básicos de un calorímetro. En este caso, se trata de un calorímetro de titulación isotérmica (CTI). Éste tiene dos celdas idénticas, una de referencia (R) que se llena de agua pura y otra que contiene la muestra (M).

Figura 1. El Calorímetro de Titulación Isotérmica (CTI) consta de dos celdas idénticas, la de referencia R que se llena de agua pura y la de la muestra M que se llena de una solución de proteína P. En la celda M se coloca una jeringa que contiene una solución de una sustancia L cuya interacción con la proteína se desea estudiar. El calorímetro establece una diferencia de temperatura ΔT entre ambas celdas que se encuentra aisladas del exterior por una chaqueta térmica. Ambas están colocadas dentro de una "chaqueta térmica" que las aísla del exterior, evitando así pérdida o ganancia de energía desde el entorno. El calorímetro establece una diferencia de temperatura (ΔT) muy pequeña, aproximadamente de 0.0001 grados o menos, de tal manera que la medición se realiza a temperatura constante, y de ahí el empleo de la palabra isotérmico en el nombre del calorímetro. En la celda M se coloca la punta de una jeringa que permite inyectar pequeñas cantidades de su contenido. Típicamente, en la celda M se sitúa una solución de una proteína (P) y en la jeringa una solución de una sustancia (L) cuya interacción con la proteína se desea estudiar. Al realizar una primera inyección o titulación, L y P entran en contacto dentro de la celda M e interaccionan entre sí, liberando o absorbiendo calor. Así, la celda M se calienta o se enfría y el ΔT original cambia. El CTI entonces proporciona energía a la celda que esté a menor temperatura para recuperar el ΔT original. Esto lo hace a través de unos dispositivos llamados termopares. De hecho, lo que el CTI realmente mide no es el cambio de temperatura ΔT sino la potencia (corriente y voltaje) proporcionada a través de estos dispositivos. La cantidad de energía que hay que proporcionar para recobrar el ΔT original es igual a la cantidad de energía que se liberó o absorbió debido a la interacción o unión entre P y L en esa primera inyección. Esta energía es el área del primer pico de la Figura 2. En subsecuentes titulaciones, la cantidad de energía debida a la unión de L a P disminuye, ya que la cantidad de P libre en la celda (sin tener a L unido) es cada vez menor (Figura 2). Es decir, las áreas de los picos van decreciendo. En un experimento exitoso, en las últimas titulaciones esta energía es muy cercana a cero, ya que todas las moléculas de proteína tienen unido a L. Con un CTI, entonces, la medición se realiza a temperatura constante, variando la concentración de la sustancia que esté colocada en la jeringa.
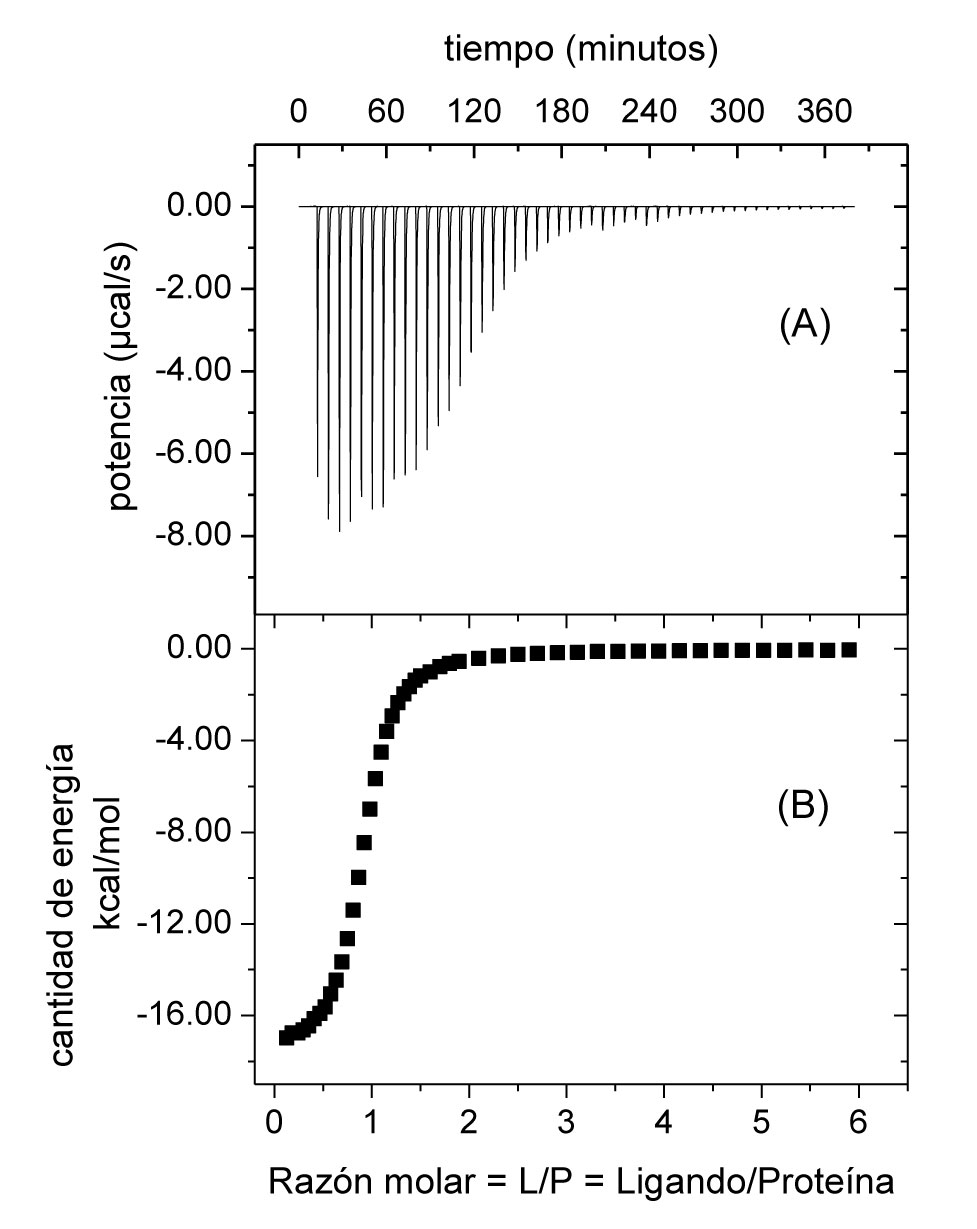
Figura 2. (A) Titulaciones sucesivas de L en la solución de proteína P; (B) cantidad de energía debida a la interacción entre L y P a diferentes razones molares L/P.
Otro instrumento que es muy útil es el calorímetro diferencial de barrido (CDB) que se ilustra en la Figura 3. En este caso, en la celda M se coloca una proteína y la celda R se llena con el mismo solvente en el que ésta se encuentra (normalmente, una solución acuosa de un amortiguador). En un experimento de CDB ambas celdas son calentadas a la misma velocidad y, como son idénticas, el ΔT es el mismo a toda temperatura hasta que, como con los CTI, ocurre algún evento que libera o absorbe energía en la celda M. En el CDB se mide entonces la concentración constante de la muestra en la celda M, variando la temperatura. Los equipos modernos son capaces de calentar desde velocidades muy bajas (0.2 grados cada minuto) hasta muy rápidas (4.2 grados cada minuto), y operan aplicando una presión ligera (entre 1 y 2 atmósferas) a ambas celdas para inhibir durante el calentamiento la formación de burbujas de los gases disueltos en la muestra.

Figura 3. El Calorímetro Diferencial de Barrido (CDB) consta de dos celdas idénticas, la de la muestra M que se llena de una solución de proteína P, y la de referencia R que se llena de agua pura o con el mismo solvente en el que está la proteína.
Estos calorímetros son muy útiles para estudiar lo que le ocurre a una proteína cuando ésta se calienta: el complejo arreglo tridimensional de los átomos que la forman (conocida como estructura nativa) se altera drásticamente hasta alcanzar un conjunto de estructuras desordenadas indistinguibles entre sí (conocidas como el estado desnaturalizado). Esta transición del estado nativo al desnaturalizado ocurre en un intervalo de temperaturas, que es lo que mide el CDB. La Figura 4 ilustra una de estas transiciones o traza calorimétrica (que es como una huella digital de la proteína). Dado que la estructura nativa de cada proteína es distinta, la traza calorimétrica de cada una de ellas es diferente. Midiendo estas trazas con un CDB y analizando sus características a través del empleo de modelos, es posible avanzar en la comprensión de las múltiples interacciones que mantienen a una proteína en su estado o estructura nativa, estado en el que están cuando realizan sus funciones.
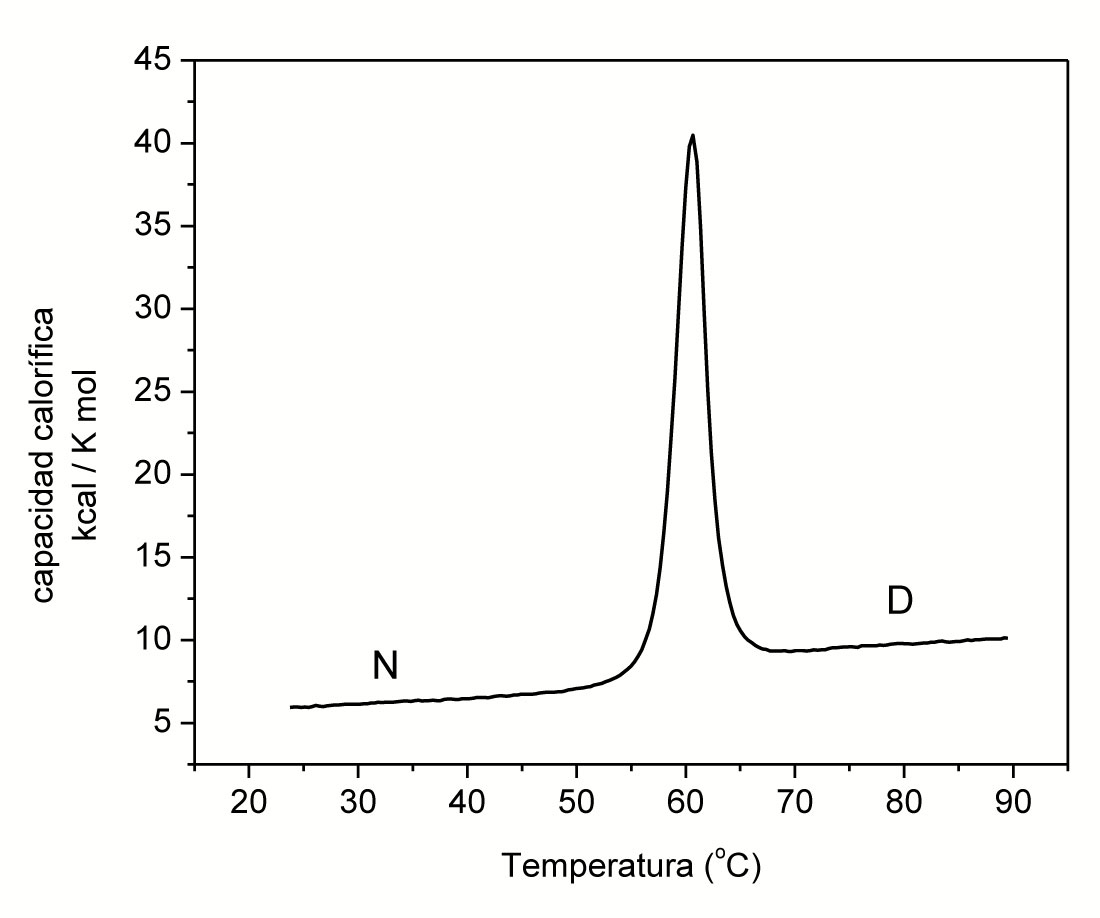
Figura 4. Traza calorimétrica de una proteína. Al calentar, la proteína pasa del estado nativo (N) a bajas temperaturas al estado desnaturalizado (D) a altas temperaturas.
Proteínas y calorimetría
A las proteínas se les conoce como las máquinas de la vida porque determinan la forma y estructura de las células y dirigen muchos de los procesos vitales. Las funciones de las proteínas son específicas de cada una de ellas. Además, hacen de todo: muchas de ellas realizan una función enzimática, actuando como biocatalizadores de las reacciones químicas del metabolismo celular; otras son hormonas, como la insulina y el glucagón, que regulan los niveles de glucosa en la sangre, o la calcitonina que regula el metabolismo del calcio; otro grupo de ellas tiene funciones defensivas, como la trombina y el fibrinógeno, que contribuyen a la formación de coágulos sanguíneos para evitar hemorragias. | A las proteínas se les conoce como las máquinas de la vida porque determinan la forma y estructura de las células y dirigen muchos de los procesos vitales. Las funciones de las proteínas son específicas de cada una de ellas. | |
 | ||
Todas las proteínas, sin importar cuál sea su papel específico, realizan su función de la misma manera: por unión selectiva a otras moléculas. Y todas llevan a cabo su función cuando están en su estructura nativa. Las interacciones entre las proteínas y otras moléculas y las interacciones inter-atómicas que mantienen estable su estado nativo, son interacciones débiles. Se llaman así para distinguirlas de los enlaces covalentes entre átomos, que son mucho más energéticos. Estas interacciones pueden romperse o establecerse con extraordinaria facilidad y son de varios tipos: puentes de hidrógeno, interacciones entre iones, interacciones hidrofóbicas y fuerzas de van der Waals. Gracias a los avances tecnológicos de los últimos años, las pequeñas cantidades de energía involucradas en la formación o ruptura de interacciones débiles pueden medirse empleando calorímetros como el CTI o el CDB, cada día con mayor precisión y usando menor cantidad de muestra.
La inhibición de la TIM de un parásito
En el año 2002, el Dr. Gómez Puyou estaba interesado en encontrar sustancias que fueran capaces de inhibir la función de una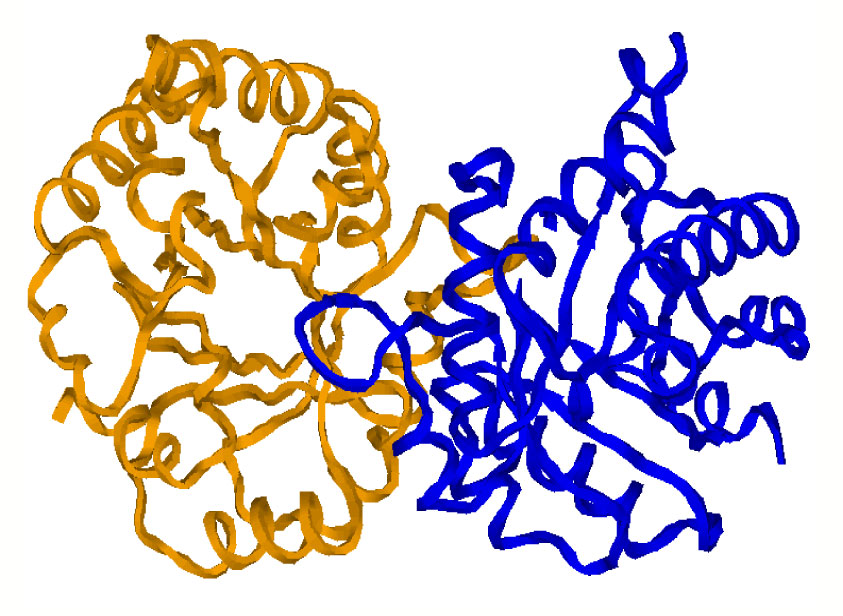
Figura 5. Proteína Triosa Fosfafo Isomerasa (TIM) en su estado nativo. Esta proteína es un homodímero formado por dos monómeros idénticos (en colores amarillo y azul, respectivamente), cada uno con 250 amino ácidos. En esta representación sólo se muestra la cadena principal, a la cual están unidos los amino ácidos. proteína llamada triosa fosfato isomerasa (TIM). La TIM es una enzima (Figura 5) que interviene en uno de los pasos de la glucólisis, una vía metabólica universal por la cual las células obtienen energía. Su intención era inhibir a la TIM de un parásito (Trypanosoma cruzi) que causa la enfermedad conocida como Mal de Chagas. La idea era que el inhibidor –que eventualmente podría convertirse en un fármaco– inhibiera a la TIM del parásito que entonces moriría de inanición, al ser incapaz de obtener energía. Esto debería llegar a ocurrir en el paciente infectado, y por tanto se requería que el inhibidor fuese inactivo frente a la TIM de humano. Es decir, esta estrategia de búsqueda de una solución al Mal de Chagas demandaba una gran especificidad: un inhibidor que fuese tal para la TIM del parásito pero fuese inocuo frente a la TIM de humano.
El Dr. Gómez Puyou, sus colaboradores y sus alumnos de posgrado, habían encontrado varios inhibidores que tenían esas características. Todos ellos eran sustancias conocidas como benzotiazoles. El más efectivo de ellos era un compuesto cuyo nombre formal es 3-(2-benzotiazoliltio) 1-propansulfonato de sodio (Figura 6), pero al que nos referíamos por "compuesto 8" por ser el octavo de una lista de posibles inhibidores.
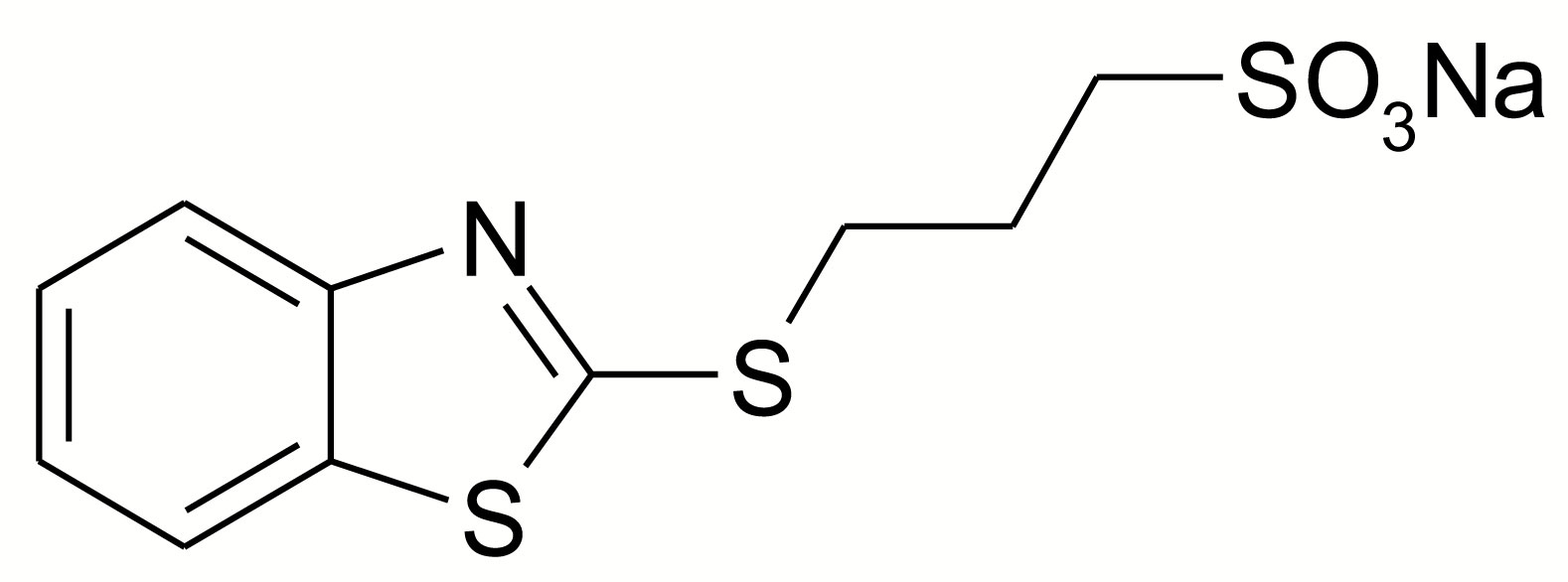
FIGURA 6. Inhibidor de la actividad catalítica de la TIM. El nombre químico de este benzotiazol es 3-(2-benzotiazoliltio) 1-propansulfonato de sodio. Este compuesto posee una geometría molecular que tiene dos anillos planos e hidrofóbicos unidos, una corta cadena hidrofóbica flexible y un grupo cargado en un extremo. Así, aquella tarde decidimos que para contestar ¿cómo se une el compuesto 8 a la TIM de Trypanosoma cruzi (TcTIM)? y ¿cuántas moléculas del compuesto 8 se unen a cada TcTIM? podríamos emplear un CTI.
Después de varios experimentos preliminares para encontrar las concentraciones óptimas para realizar la medición, pusimos la TcTIM en la celda a 0.1 mM y el compuesto 8 en la jeringa a 10 mM, ambos disueltos en el mismo amortiguador, y realizamos las titulaciones. El resultado (Figura 7) fue que a medida que el compuesto 8 se fue acumulando en la celda, la señal cambió de signo, es decir, la celda M se enfrió en las primeras titulaciones y se calentó en las subsecuentes. Se concluyó que la TcTIM tiene 2 sitios diferentes en su estructura a los cuales se une el compuesto 8 (una molécula de compuesto 8 en cada sitio). El experimento de CTI también expresó que la unión al primer sitio es endotérmica, es decir, se absorbe energía al ocurrir, mientras que la unión al segundo sitio es exotérmica, esto es, se libera energía al suceder. En el lenguaje de la termodinámica, este hallazgo se expresa diciendo que la primera unión es controlada entrópicamente y la segunda entálpicamente. La implicación es que para la unión al primer sitio la geometría molecular del compuesto 8 es crucial, mientras que la unión al segundo sitio está determinada por las interacciones entre los diversos grupos químicos del compuesto 8 y los amino ácidos de la TcTIM que están en sus sitios de unión.
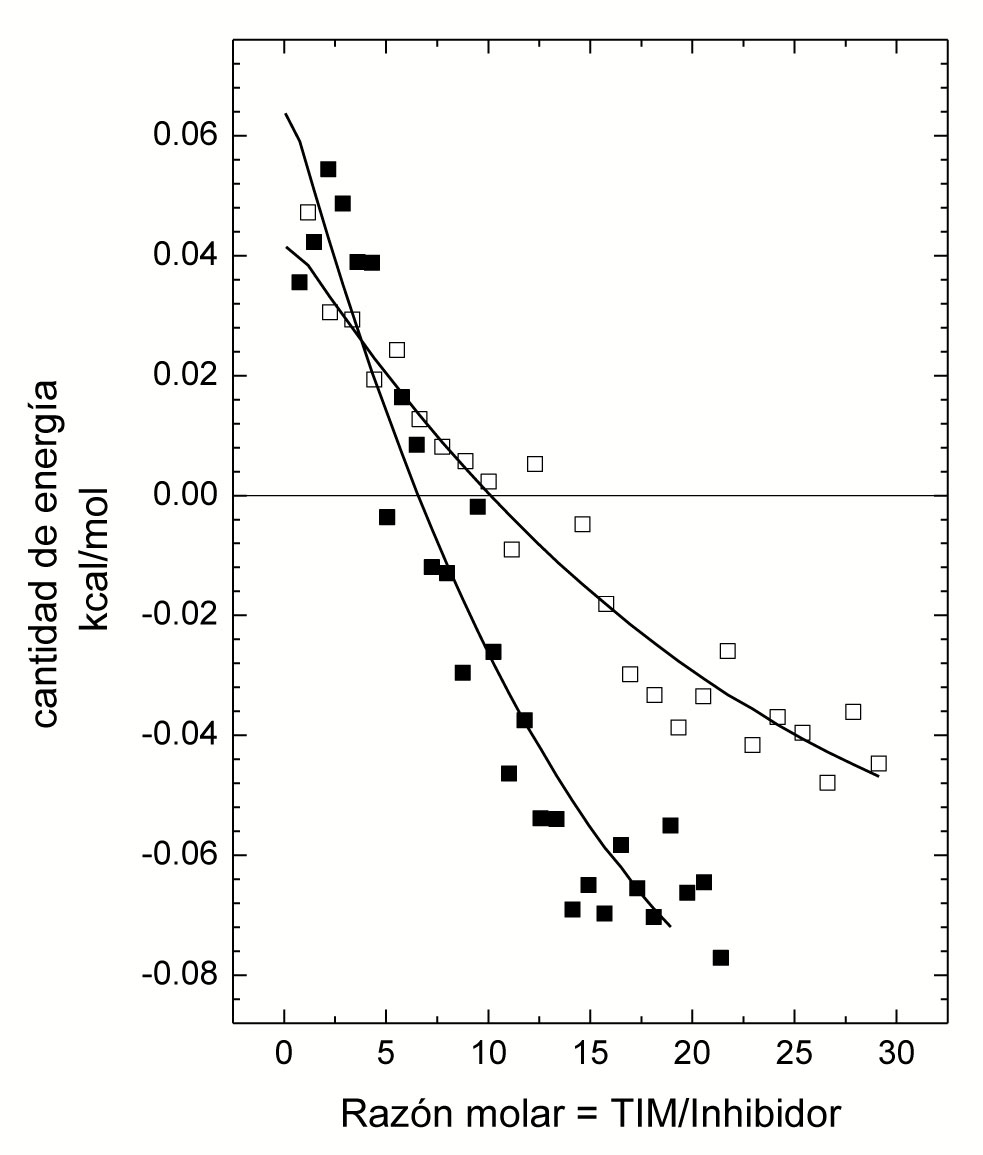
Figura 7. Resultado de un experimento de CTI empleando la proteína TIM y el benzotiazol en la Figura 6. Los símbolos abiertos y cerrados son para dos concentraciones diferentes de TIM (TÉLLEZ-VALENCIA et al., 2004).
El compuesto 8 nunca llegará a ser un fármaco contra el Mal de Chagas. La razón principal es que la concentración necesaria para inhibir efectivamente a la TcTIM es mayor a la máxima concentración recomendada para que una sustancia pueda ser empleada como fármaco. Entonces, más allá de comprender mejor la unión del compuesto 8 a la TcTIM, ¿para qué sirvió este estudio calorímetrico? La respuesta es que proporcionó información valiosa que favoreció el diseño en el pizarrón y la síntesis en el laboratorio de otros benzotiazoles que resultaron ser mucho mejores inhibidores de la TcTIM (más rápidos y a menor concentración) que el compuesto 8. El problema que se enfrenta ahora es el de encontrar la manera de hacer llegar estos compuestos al interior del parásito, atravesando su membrana exterior.
La desnaturalización térmica de la TIM de humano
Algunos años después, decidimos que sería relevante hacer un estudio sistemático de la desnaturalización térmica de la TIM de varias especies, especialmente de diversos parásitos y de la de humano. Las proteínas se obtuvieron de manera recombinante en bacterias (se usó Escherichia coli), un procedimiento muy común para facilitar su purificación y aumentar rendimiento. El objetivo central era llegar a comprender a nivel molecular las diferencias entre las características de la desnaturalización de varias TIMs, es decir, de proteínas cuya secuencia de amino ácidos es diferente pero que, dado que realizan la misma función, se denominan con el mismo nombre. Para este estudio, se decidió emplear la CDB y comenzar a medir muestras de varias TIMs. Encontramos lo esperado: trazas calorimétricas como las de la Figura 4 donde el área bajo la traza, su ancho y la temperatura a la cual ocurre el máximo era diferente para cada TIM. Mientras se acumulaban esos datos, "saltó la liebre": para la TIM de humano (HsTIM), en lugar de aparecer un solo pico en la traza calorimétrica, ¡aparecieron dos! (Figura 8). Llegar a comprender este hallazgo se convirtió en una prioridad (obsesión es un mejor calificativo) que llevó tres años y nos desvió –sólo temporalmente– del objetivo inicial, pero logramos entender, por el momento al menos, porque en ciencia hay pocos resultados que sean realmente definitivos.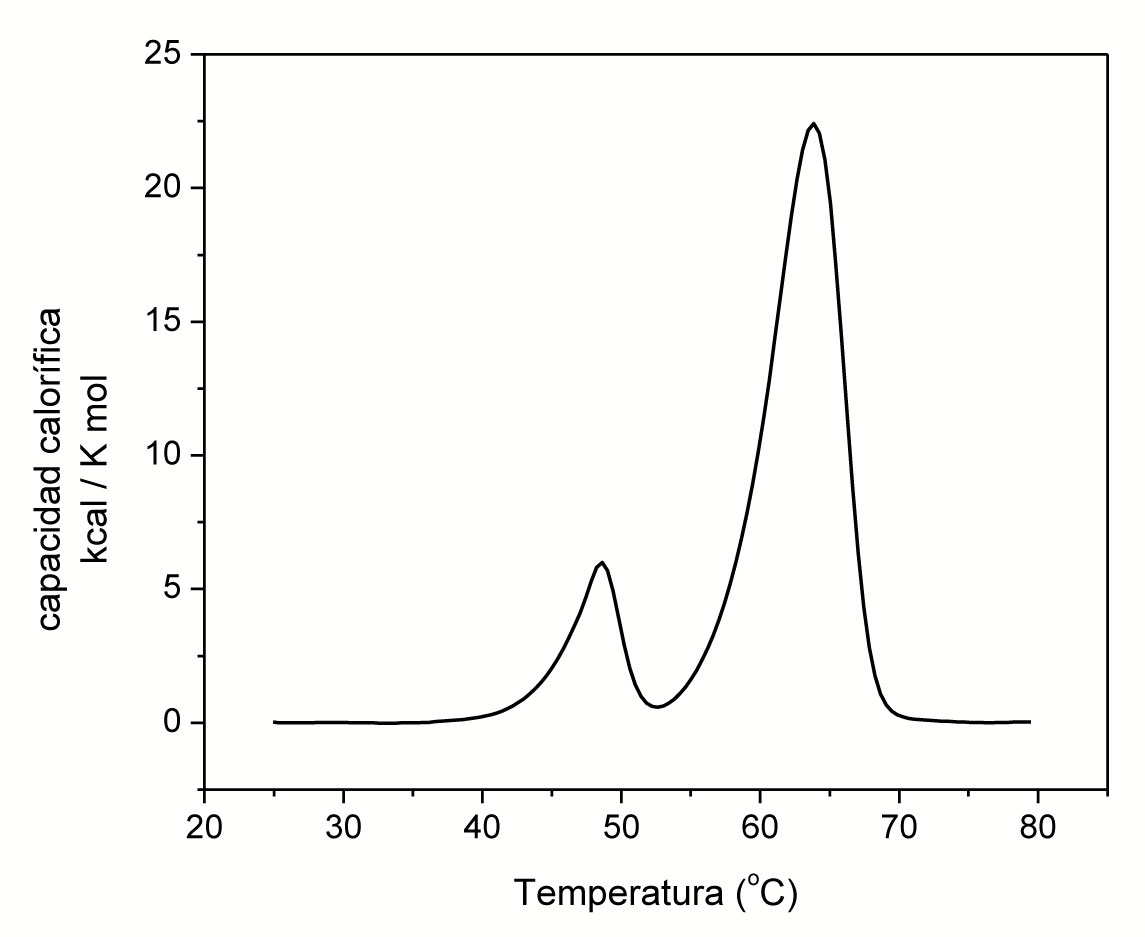
Figura 8. Resultado de un experimento de CDB empleando la proteína TIM de humano (AGUIRRE et al., 2011).
La TIM es un homodímero, formado por dos monómeros idénticos, cada uno con 250 amino ácidos (Figura 5). Los monómeros están adheridos uno al otro sólo mediante interacciones débiles, sin que haya entre ellos ningún enlace covalente. Después de descartar que la muestra de HsTIM estuviese contaminada con alguna otra proteína diferente a la TIM, la siguiente hipótesis para explicar la traza con dos picos fue postular que el pico de baja temperatura se debía a la disociación del dímero en monómeros, y el segundo a la desnaturalización de los monómeros. Esta hipótesis resultó falsa, ya que al lograr separar las poblaciones de proteína correspondientes a cada pico, se determinó que ambas proteínas tenían actividad de TIM. Es decir, ¡estábamos ante la presencia de dos HsTIM con estabilidades térmicas muy diferentes! Las preguntas a contestar fueron entonces ¿cuál es la diferencia entre ellas? y ¿qué la causó?
La respuesta al primer cuestionamiento es que la proteína que se desnaturaliza a baja temperatura tiene un peso molecular mayor en 28 daltones, una diferencia muy sutil ya que corresponde a sólo el 0.05% del peso molecular total de la proteína (que es de 54 000 daltones). La diferencia entre ambas TIMs se encuentra en el amino ácido que ocupa la posición 99 de uno de los monómeros: en la proteína que produce el pico de baja temperatura existe una arginina, mientras que en el de alta temperatura una lisina ocupa ese lugar. La causa de esta sustitución es que la maquinaria (el ribosoma) que fabrica la HsTIM en Escherichia coli comete un error al sintetizar algunos de los monómeros. Así que el primer pico observado en el experimento de CDB es producido por la desnaturalización de una HsTIM que es un heterodímero (en uno de monómeros hay una arginina y en el otro una lisina), y el segundo pico por una HsTIM que es un homodímero (ambos mónomeros realmente idénticos con lisina en sus posiciones 99). La falla del ribosoma puede corregirse y, cuando esto se hace, se produce una sola HsTIM con lisina en la posición 99 en ambos monómeros, que tiene un solo pico en su traza calorimétrica.
El empleo del CDB fue crucial en este proyecto. Sin él, hubiese sido prácticamente imposible descubrir que teníamos dos HsTIM con estabilidades térmicas muy diferentes. En la actualidad seguimos estudiando la formación homo y heterodímeros de TIMs, cuyos monómeros difieren en el amino ácido que está en la posición 99. Sin duda, para llevar a buen término este nuevo proyecto nos hará mucha falta contar con el talento y entusiasmo del Dr. Gómez Puyou. Lo extrañamos.
Bibliografía
AGUIRRE, B. "A ribosomal misincorporation of Lys for Arg in human triosephosphate isomerase expressed in Escherichia coli gives rise to two protein populations". PLoS ONE, 2011, 6(6): e21035.COOPER, Alan. Tutorial Chemistry Texts, The Royal Society of Chemistry, 2004.
TÉLLEZ-VALENCIA et al. "Inactivation of triosephosphate isomerase from Trypanosoma cruzi by an agent that perturbs its dimer interface". J. Mol. Biol., 2004, 341 (5), 1355-1365.
COMENTARIOS
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO
Dirección General de Cómputo y de Tecnologías de Información y Comunicación
Dirección General de Cómputo y de Tecnologías de Información y Comunicación
2013 Esta obra está bajo una licencia de Creative Commons