|
En mamíferos, debido al menor tamaño de las poblaciones
efectivas, se considera que los sitios sinónimos no se encuentran
bajo presiones selectivas significativas. Las especies de mamíferos
también presentan sesgo en el uso de codones sinónimos (Eyrewalker
1991; Ikemura and Wada 1991). Sin embargo, los sesgos se han atribuido
a las variaciones en concentraciones de nucleótidos en el genoma
(i.e. isocoros: (Bernardi 1995; Bernardi et al. 1997). Estas variaciones
son, aparentemente, particulares a los organismos que autorregulan su
temperatura. (Ver. (Hughes et al. 1999). Debido a que ciertas regiones
en el genoma son más ricas en guanina y citosina (GC), los genes
en estas regiones muestran una mayor preferencia hacia los codones que
terminan en G o C. En regiones ricas en adenina y timina (AT), ocurre
el caso contrario. De este modo, la composición local de bases
induce un sesgo en el uso de codones. Pero, ¿todo el sesgo en la
distribución de codones sinónimos es producto de las variaciones
en las concentraciones de nucleótidos?
 Con
el objetivo investigar si el sesgo en el uso de codones (SUC) observado
en mamíferos puede explicarse completamente por el sesgo en el
uso de nucleótidos y en caso contrario, investigar qué variables
podrían estar implicadas en el SUC residual observado, hemos analizado
(Urrutia y Hurst, 2001) el uso de codones alternativos en una muestra
de 2396 secuencias de genes humanos. Inicialmente analizamos si el uso
de codones observado en los genes humanos podía explicarse por
las distribuciones de nucleótidos. Para ello realizamos tres análisis
contrastando las preferencias observadas, con las expectativas derivadas
de las concentraciones de nucleótidos. Encontramos que 81% de los
genes presentaban un sesgo superior al esperado de acuerdo a la distribución
de las secuencias aleatorias indicando que los sesgos observados en la
mayoría de los genes no pueden explicarse por los niveles básales
de nucleótidos. Adicionalmente, encontramos preferencias sistemáticas
en los genes por ciertos codones (Urrutia y Hurst, 2001).
Con
el objetivo investigar si el sesgo en el uso de codones (SUC) observado
en mamíferos puede explicarse completamente por el sesgo en el
uso de nucleótidos y en caso contrario, investigar qué variables
podrían estar implicadas en el SUC residual observado, hemos analizado
(Urrutia y Hurst, 2001) el uso de codones alternativos en una muestra
de 2396 secuencias de genes humanos. Inicialmente analizamos si el uso
de codones observado en los genes humanos podía explicarse por
las distribuciones de nucleótidos. Para ello realizamos tres análisis
contrastando las preferencias observadas, con las expectativas derivadas
de las concentraciones de nucleótidos. Encontramos que 81% de los
genes presentaban un sesgo superior al esperado de acuerdo a la distribución
de las secuencias aleatorias indicando que los sesgos observados en la
mayoría de los genes no pueden explicarse por los niveles básales
de nucleótidos. Adicionalmente, encontramos preferencias sistemáticas
en los genes por ciertos codones (Urrutia y Hurst, 2001).
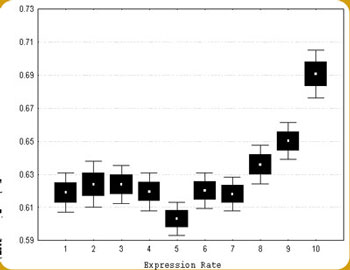
Si el uso de codones particulares incrementa
la eficiencia en la síntesis
de proteínas, entonces se puede esperar que los genes de mayor actividad
y por lo tanto requieran mayor eficiencia, tendrán un sesgo mayor
en el uso de codones. Se utilizó un método novedoso (Urrutia
and Hurst 2001) para medir el sesgo en el uso de codones corrigiendo las
variaciones entre proporciones de nucleótidos, en mas de 8000 genes
humanos. Dicho método implica el cálculo de frecuencias esperadas
de codones para cada aminoácido con base en la frecuencia de uso
de nucleótidos en los sitios sinónimos de los otros aminoácidos.
De este modo, se consigue corregir el impacto de las variaciones locales
de la composición de nucleótidos. Los resultados mostraron
una relación entre los niveles de uso preferencial de codones con
los niveles de expresión, los genes con mayor actividad presentaban
mayores sesgos en el uso de codones (Urrutia and Hurst 2003).
Estos resultados sugieren que, a pesar
de que las proporciones en las que se usan los codones alternativos
están determinadas en gran medida
por variaciones en las concentraciones de nucleótidos a lo largo
del genoma, las concentraciones de nucleótidos observadas no son
suficientes para explicar los sesgos en el uso de codones en los genes
humanos. La correlación entre los niveles de actividad génica
y el nivel de sesgo en el uso de codones sugieren la existencia de presiones
selectivas significativas actuando sobre la elección de codones
presumiblemente relacionadas con la eficiencia de la síntesis de
proteínas.
|
