El Misterio de los Intrones Palabras Clave : intrones, computación evolutiva, algoritmos genéticos, programación genética, .
Los intrones o segmentos de ADN no codificadores, son igualmente misteriosos en computación evolutiva que en Biología, pareciendo desafiar a la teoría misma de la evolución. En este artículo se habla sobre los intrones en computación evolutiva y su analogía con sus símiles biológicos, indicando además algunos de sus posibles efectos positivos y negativos, así como las rutas de investigación más promisorias dentro de este intrigante tema.
Recientemente ha habido un interés notable por parte de la comunidad de Computación Evolutiva, en torno al estudio de los segmentos de una cadena cromosómica que no contribuyen a su valor de aptitud, pero que están presentes durante el proceso evolutivo. A estos segmentos se les denomina "intrones" y parecen tener extraordinarias semejanzas con sus símiles naturales, los cuales tampoco han podido ser explicados de manera satisfactoria por los biólogos. Los intrones biológicos (o ADN [a] de desecho) son "segmentos no codificadores" de ADN (es decir, no se involucran en la codificación para producir proteínas), pero se cree que tienen alguna función que permanece desconocida por los biólogos. Los intrones son una consecuencia natural del proceso evolutivo en la programación genética [1], y han sido inducidos artificialmente también en los algoritmos genéticos [8a] Tanto los estudios teóricos, como la experimentación realizada hasta ahora, parecen sugerir que la presencia de los intrones podría motivar la recombinación y la destrucción de ciertos bloques constructores existentes en los algoritmos evolutivos. La evidencia existente parece indicar que los intrones tienen un efecto estabilizador, mejorando la capacidad de los algoritmos evolutivos para preservar los buenos bloques constructores, lo cual es una característica altamente deseable de tales algoritmos. En este artículo daremos una visión general de este interesante tema, con la explicación de algunas similitudes entre los intrones biológicos y los simulados, así como una revisión del trabajo más relevante al respecto, desde la perspectiva de la computación evolutiva. Finalmente, se hablará sobre algunas de las rutas promisorias de investigación en relación con el tema. Antecedentes Dada la estrecha relación de esta área con la biología, resulta necesario comenzar por explicar un poco la terminología que será de utilidad para entender mejor el contenido de este artículo. Comenzaremos por hablar de los dos tipos de células que conforman a los seres vivos: eucariotas y procariotas. Las células eucariotas tienen organelos y un núcleo, ambos cubiertos por una membrana. El núcleo contiene el material genético de la célula en este caso. Las células procariotas no tienen una membrana alrededor del núcleo, ni alrededor de sus organelos, y almacenan su material genético en una sola molécula grande de ADN. Todos los organismos procariotas son unicelulares, mientras que los eucariotas pueden tener una o varias células [2a] Las proteínas, que se consideran los bloques constructores de la vida, son el tipo de molécula orgánica más abundante en los seres vivos. Una proteína consta de una o más cadenas polipéptidas, es decir, cadenas de amino-ácidos. Un amino-ácido es una molécula orgánica que consta de un átomo de carbono, unido a un átomo de hidrógeno, a un grupo carboxilo, a un grupo amino y a un grupo que varía según el amino-ácido de que se trate. Hay 20 diferentes amino-ácidos considerados importantes en genética. El orden de los amino-ácidos en las cadenas polipéptidas y la estructura de dichas cadenas, son lo que le proporciona a las proteínas sus capacidades estructurales o funcionales. Cuando un organismo se reproduce, es imperativo que las instrucciones para construir sus proteínas se reproduzcan de manera precisa y completa. Estas instrucciones son mantenidas por un segundo tipo de molécula orgánica, a la que se denomina nucleótido. Los nucleótidos se unen para formar moléculas de mayor tamaño a las que se denomina ácidos nucleicos. Los dos tipos más comunes de ácidos nucleiclos son el ADN (ácido desoxirribonucleico) y el ARN (ácido ribonucleico). En los organismos eucariotas, el ADN se combina con las proteínas para formar cromosomas, los cuales se localizan en el núcleo de una célula. El conjunto completo de cromosomas de un organismo se denomina genoma. El ADN es el material genético que se propaga de generación en generación y contiene las instrucciones sobre cómo construir las proteínas necesarias para un organismo en particular. Aunque toda la información genética se almacena en el ordenamiento de los nucleótidos en el ADN, éste no está involucrado directamente en la síntesis de proteínas. El ADN dirige la síntesis de proteínas enviando instrucciones en forma de ARN, el cual es el encargado de llevar a cabo la tarea. Un
gene es un segmento de ADN, que se codifica para un producto de ARN.
A los diferentes valores de un gene se les llama alelos.
Figura 1 : Los genes se encuentran delimitados por un sitio de inicio y un sitio de terminación.
La síntesis de proteínas, a partir del ADN, ocurre en dos pasos, a los que se denomina transcripción y traducción. Durante la transcripción, el ADN de un gene se copia al ARN. Sólo se transcribe un segmento de ADN en un cromosoma. Un gene es marcado con un sitio de iniciación y otro de terminación. Los sitios de iniciación contienen una o más regiones reguladoras y una región promotora (ver figura 1). Las regiones reguladoras inhiben o permiten la expresión de un gene. Las regiones promotoras son reconocidas por una enzima llamada polimerasa ARN, como puntos de inicio para la transcripción. La transcripción del gene continúa hasta que la polimerasa ARN encuentra el sitio de terminación. En este punto, se concluye la transcripción y se libera el ARN transcrito y la polimerasa ARN. Hay varios tipos de productos de ARN, de los cuales 3 (el ARN mensajero o ARNm, el ARN de transferencia o ARNt y el ARN ribosomal o ARNr) tienen funciones específicas durante la traducción de la síntesis de proteínas. Durante el proceso de traducción, el ARNm, el ARNt y los ribosomas (que están hechos de ARNr y proteínas), trabajan juntos para construir una proteína. El ARNm contiene el ordenamiento adecuado de los amino-ácidos, tal y como se copió del ADN. Este ordenamiento está almacenado en la forma de codones, que son tercetos de nucleótidos que representan, ya sea a un amino-ácido o a una señal de terminación. El ARNt "lee" el ARNm a razón de 3 nucleótidos a la vez, recuperando el amino-ácido correcto del citoplasma. El ribosoma se une al ARNm y se mueve secuencialmente hacia la parte inferior de la cadena. Conforme el ARNt recupera amino-ácidos en el orden correcto, el ribosoma une cada nuevo amino-ácido a la cadena polipéptida. Cuando se alcanza el final de la cadena de ARNm, el ribosoma se separa del ARNm y libera una cadena polipéptida completa [2b] ADN no codificador El término ADN no codificador se refiere a todo el ADN que no está involucrado en la codificación de un producto maduro de ARN. Aunque el ADN no codificador es común en los sistemas biológicos, su origen y funciones sigue siendo un misterio. Existen tres tipos de ADN no codificador: las regiones intergenéticas, las regiones intragenéticas y los pseudogenes Aunque los genes yacen linealmente a lo largo de los cromosomas, no se encuentran en posiciones necesariamente contiguas. Las regiones intragenéticas son las porciones de ADN que se encuentran entre los genes. Estas regiones no se transcriben a ARN. Se sabe que algunas porciones de las regiones intragenéticas regulan la expresión de los genes adyacentes a ellas, pero otras no tienen ninguna función conocida. Las regiones intragenéticas, llamadas también intrones, son segmentos de ADN encontrados dentro de los genes. Los intrones se transcriben a ARN junto con el resto del gene, pero deben ser removidos del ARN antes de que se complete el producto maduro de ARN. El ARN que todavía contiene regiones intrónicas es llamado pre-ARN. Después que se le remueven los intrones, a los segmentos restantes de ARN se les denomina exones [b] o (regiones expresadas), y se les une para transformarse en un producto maduro de ARN. Todo esto debe ocurrir con una precisión asombrosa. Si el corte o la unión del ARN está fuera de lugar una sola posición, el error producido arruina toda la proteína. Un pseudogene es un segmento de ADN que es similar a un gene funcional, pero que contiene cambios de nucleótido para prevenir su transcripción o traducción. Se cree que los pseudogenes se originan en la duplicación genética o en la transcripción inversa de ARN. Un detalle interesante es que los pseudogenes producidos de una transcripción inversa no contienen intrones. Puesto que los pseudogenes no están expresados, no están sujetos a la presión de selección del ambiente. Por lo tanto, los pseudogenes acumulan mutaciones rápidamente. Cuando los pseudogenes se mutan lo suficiente como para que su similitud con un gene funcional no resulte aparente, se vuelve simplemente ADN intergenético no codificador.
Figura
2 : Ejemplo de la presencia de intrones
en el ADN. I Los intrones biológicos fueron descubiertos en 1977, poniendo de cabeza a la comunidad científica que no pudo predecir su aparición a partir del paradigma neo-Darwiniano de la evolución [c] . Los intrones se encuentran solamente en los organismos eucarióticos, en los cuales constituyen una enorme porción de sus genomas. Por ejemplo, alrededor del 30% del genoma humano está formado por intrones. Sólo alrededor del 3% consiste en ADN codificador y el resto del genoma consiste en ADN no codificador, secuencias repetitivas y regiones regulatorias. La colocación inusual de los intrones, interrumpiendo las regiones codificadoras de los genes [d](ver figura 2), y el hecho de que se requiere energía extra para mantener y procesar estas estructuras que no parecen tener ninguna función, ha hecho de los intrones uno de los temas de investigación más intrigantes en Biología. Su mera existencia es un desafío para la teoría de la evolución, ya que si contribuyeran negativamente al proceso genético, la selección natural debió haberlos eliminado desde hace mucho tiempo. Para Richard Dawkins [5], los intrones son el más puro ejemplo del "ADN egoísta", que está ahí simplemente para su propia supervivencia. La investigación realizada hasta ahora en torno a los intrones, se ha centrado en responder a 3 preguntas:
De estas 3 preguntas, la primera es la que tiene la respuesta más satisfactoria. Se sabe que los intrones son removidos del ARN mediante un proceso denominado empalme del ARN, el cual es una especie de "edición" del contenido del ARN (es decir, es un proceso de remoción de intrones), que ocurre dentro del núcleo de una célula. Existen diferentes métodos de empalme del ARN, y la mayoría requiere de la ayuda de proteínas que reconocen secuencias específicas en el pre-ARN. La segunda pregunta sigue siendo tema de estudio. La denominada "teoría exónica de los genes" [6] sugiere que los exones son los bloques constructores de las proteínas y que los genes se crean a partir de las combinaciones de estos bloques constructores. Esta teoría nos lleva hacia la "hipótesis de barajeo de exones", [3] la cual afirma que los intrones incrementan el porcentaje de recombinación de los exones, haciendo más fácil moverlos y crear nuevos genes. Walter Gilbert afirma que [7]: "...los intrones [estadísticamente hablando] representan puntos de gran propensión a la recombinación: por su mera presencia y longitud, incrementan el porcentaje de recombinación, y en consecuencia incrementan el barajeo de exones por factores del orden de 106 a 108". Hay evidencia que parece sugerir que los exones pueden corresponder a sub-unidades estructurales y funcionales de las proteínas, incluyendo ejemplos específicos del mismo exón, que existe en diferentes genes donde se requiere la misma estructura o funcionamiento de dos proteínas distintas. De acuerdo a esta teoría, la estructura intrón-exón de los genes eucarióticos, motiva la formación de nuevos genes a partir de sub-unidades estructurales y funcionales de los genes existentes, en lo que constituye un proceso más eficiente que construir los nuevos genes a razón de un nucleótido a la vez. Ante esta teoría, la pregunta que resulta más obvia es: si los intrones son útiles para la recombinación, entonces ¿por qué sólo aparecen en los organismos eucariotas y no en los procariotas? Respecto a la tercera pregunta, existen dos teorías comúnmente aceptadas: la teoría de los "intrones tempranos" y la de los "intrones tardíos". Según la primera, los ancestros de los eucariotas y los procariotas poseían intrones, pero éstos últimos los perdieron durante su proceso evolutivo. De acuerdo con la segunda teoría, estos ancestros no poseían intrones y los eucariotas los obtuvieron durante el proceso evolutivo. La teoría de los "intrones tempranos" sugiere que el último ancestro común de los procariotas y los eucariotas, tenía intrones en su genoma. Para dar cabida a sus cortos períodos reproductivos y de vida, los procariotas, subsecuentemente, perdieron los intrones de sus genomas debido a la selección natural que buscó incrementar la eficiencia de la expresión de sus genes, así como una reducción en el tamaño de sus genomas. Según esta teoría, el precio que hubieron de pagar los organismos procariotas por esta mayor eficiencia, fue el decremento en el potencial para evolucionar más en el futuro, ya que se perdieron los intrones que podrían ayudar al barajeo de exones. Los organismos eucariotas, por su parte, continuaron evolucionando con la asistencia de sus intrones y dieron lugar a formas de vida mucho más complejas. De ahí que haya mucho menos complejidad y variación en los organismos procariotas que en los eucariotas. La teoría de los "intrones tardíos" sugiere que éstos se desarrollaron en el proceso evolutivo eucariótico. Puesto que los organismos procariotas han sido considerados tradicionalmente más primitivos que los eucariotas, el genoma aún más primitivo del ancestro común de estos dos tipos de organismos, se supone que asemejaría a un genoma procariota en lo referente a su fuerte organización. Según esta teoría, los intrones habrían sido insertados en los genomas eucarióticos, poco después de haberse originado la división entre los dos tipos de organismos (eucariotas y procariotas). Ambas teorías han sido debatidas y tienen puntos a favor y en contra. El paradigma Neo-Darwiniano todavía no ha llegado a un consenso respecto a este asunto, y existe actualmente un acalorado debate en torno a estas dos posturas.
Figura
3 :
Ejemplo de una cadena cromosómica (como las usadas en los algoritmos
genéticos) que contiene intrones. Los intrones se marcan con
una ´l´, y no producen nada al decodificarse (es decir,
al convertirse a valores decimales), si no que simplemente se ignoran.
En este ejemplo, las variables X1 y X2 codificadas en la cadena binaria
tienen, ambas, 8 bits. Nótese cómo los intrones pueden
aparecer en posiciones aisladas o consecutivas, y que pueden irrumpir
o no al conjunto de bits que conforman una variable codificada. De manera análoga al debate sostenido por la comunidad de biólogos, la comunidad de computación evolutiva tiene varias posturas respecto a los efectos benéficos y negativos que podrían tener los intrones en los algoritmos evolutivos. El primer experimento con intrones reportado en la literatura especializada, es el de Levenick, [8b]quien insertó intrones en los cromosomas de un algoritmo genético (AG) para estudiar su efecto (ver figura 3 para un ejemplo de una cadena cromosómica como las usadas con los AGs, la cual contiene intrones [e] ). Levenick reportó mejoras en el desempeño de los AGs con intrones, con respecto a los que no los usaron. Sus experimentos fueron después reproducidos y extendidos por Annie S. Wu y Robert K. Lindsey, [9] quienes concluyeron que los intrones resultan más benéficos en un AG cuando la aptitud se incrementa notablemente tras descubrirse un nuevo nivel en la etapa de búsqueda, de lo que se concluyó que los intrones reducen el efecto de la "correlación espuria " [g]. Asimismo, dedujeron que los intrones hacen que un AG sea más estable, pues su presencia hace más difícil la pérdida de "bloques constructores", una vez que éstos han sido localizados, si bien también dificultan que el AG pueda generar buenos bloques constructores con el operador de cruza, requiriendo muy probablemente que se efectúen corridas más largas para hacer converger al AG. 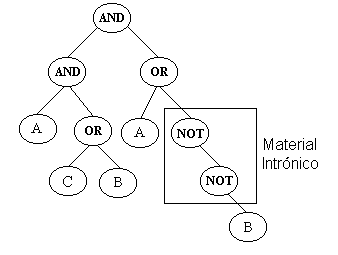
Figura 4 : Ejemplo de un árbol (como los usados en programación genética) que contiene material intrónico. Si se remueve el segmento de árbol encerrado en un cuadro, la expresión codificada tendría exactamente el mismo valor de aptitud. El primero en notar la presencia de intrones en programación genética fue Peter Angeline, quien advirtió en 1994 [10] que los árboles que John Koza hacía evolucionar en su libro de 1992, [11] contenían varios segmentos que, de ser removidos, no afectaban en lo más mínimo el valor de aptitud de esos individuos (ver figura 4 para un ejemplo de un árbol de los usados en programación genética que contiene intrones). También en 1994, Tackett [14] advirtió que los árboles evolucionados en programación genética tienden a "hincharse" (o saturarse), es decir, tienden a crecer desmesuradamente hasta alcanzar la máxima profundidad permisible. Tackett especuló que este fenómeno se debía a la presencia de segmentos de código en ciertos individuos que, aunque por sí solos no eran muy aptos, sí muy similares a segmentos de código que sí tenían una aptitud elevada. Estos bloques son también un ejemplo de "correlación espuria", como la que se presenta en los algoritmos genéticos. En investigación posterior se determinó que esta "hinchazón" de los árboles usados por la programación genética, es producida por los intrones. También se ha establecido que, contrario a lo que se creía en un principio, los intrones no son un fenómeno ocasional y esporádico, sino que llegan a constituir, en algunos casos, entre el 40% y el 60% de todo el código utilizado en etapas intermedias del proceso evolutivo en la programación genética. En etapas más avanzadas del proceso evolutivo, los intrones tienden a crecer exponencialmente y pasan a constituir la gran mayoría del código de casi toda la población [15]. Por alguna razón, los intrones en programación genética tienden a ser favorecidos por el proceso de selección. El papel que juegan, entonces, los intrones en la programación genética, es tan misterioso como el de sus símiles biológicos. Mientras que varios investigadores han determinado teórica y experimentalmente que los intrones parecen servir para proteger a los buenos bloques constructores de los efectos destructores de la cruza, su crecimiento exponencial puede producir también "estancamiento", es decir, hacer que el proceso evolutivo se detenga. De manera genérica, podemos definir a un intrón como un segmento de código (o cadena cromosómica) que no contribuye directamente al valor de aptitud de un individuo. Banzhaf et al. [1] distinguen entre diferentes tipos de intrones en programación genética, de entre los que destaca la distinción entre los intrones "locales" y los "globales". Los primeros son aquellos que no contribuyen a la aptitud de un individuo, para las entradas dadas en un cierto momento del proceso evolutivo, pero pueden afectar el valor de aptitud para otro conjunto de entradas válido. Los segundos son aquellos que no contribuyen a la aptitud de un individuo, sin importar qué valores de entrada se utilicen. Mientras que en las genotipos de longitud variable (como los árboles usados en programación genética) los intrones son "emergentes" (aparecen de forma natural durante el proceso evolutivo), en los algoritmos genéticos son normalmente "artificiales" (son insertados en la cadena cromosómica). ¿Por qué surgen los intrones? Si por un momento nos concentramos sólo en los intrones emergentes de la programación genética, la primera pregunta que se antoja es la misma que se vienen haciendo los biólogos desde hace varios años: ¿por qué surgen? Debe resultar obvio que la presión de selección no debiera dar pie a la generación de intrones, debido a que éstos no tienen un efecto directo sobre la aptitud de un individuo. Sin embargo, se cree que los intrones tienen un efecto indirecto: deben afectar la probabilidad de que los descendientes de un individuo sobrevivan. Banzhaf et al. [1] llaman a esta definición modificada de aptitud, en la que se toma en cuenta la capacidad de supervivencia de la descendencia, la "aptitud efectiva", y su valor no será función solamente de la aptitud de un individuo, sino también de su capacidad para generar descendientes con elevada aptitud, ante el uso de los operadores genéticos (cruza y mutación). Se sabe que la cruza en programación genética no suele ser mejor que un operador de mutación. De tal forma, se cree que los intrones contribuyen a aminorar los efectos nocivos de la cruza en programación genética, incrementando la "aptitud efectiva" de los individuos. Crecimiento desmesurado Se sabe que en programación genética, los intrones crecen casi siempre exponencialmente hacia el final de una corrida La razón parece ser el hecho de que los intrones pueden proporcionar una protección global, muy segura contra los efectos destructivos de la cruza. De tal forma, se piensa que los intrones se propagan más hacia el final de una corrida, porque ahí es cuando los individuos alcanzan su mejor desempeño (ya sea óptimo o sub-óptimo). Por lo tanto, la estrategia de supervivencia cambia, pues en vez de intentar mejorar el desempeño (o aptitud), lo que se hace es impedir que los efectos destructivos de los operadores genéticos hagan que tal desempeño decrezca. Ahí es donde entran en juego los intrones, que aminoran los efectos destructivos de la cruza. Los efectos nocivos de los intrones Si bien las hipótesis más aceptadas sobre el origen de los intrones parecen otorgarles un efecto positivo en el proceso evolutivo, varios investigadores han reportado también algunos de sus efectos nocivos. De ellos, el más común es el "estancamiento" (es decir, la carencia de mejoras en las aptitudes de los individuos de una población), debido al crecimiento exponencial de los intrones. Este problema, sin embargo, suele ser debatible, debido al hecho de que se presenta en etapas tardías del proceso evolutivo, en las cuales, de cualquier manera, no ocurren mejoras significativas de los individuos. Otro problema que ha sido reportado en la literatura, es el de la convergencia hacia soluciones pobres. Esto parece presentarse, sobre todo, cuando se utilizan intrones artificiales (o sea, inducidos) en programación genética. Por ejemplo, Andre y Teller [17] han reportado la obtención de resultados muy pobres al agregar intrones artificiales en su sistema. Los intrones utilizados, en este caso, eran nodos que fueron diseñados deliberadamente para no hacer nada, sino simplemente consumir más espacio en el código. Algunos investigadores argumentan que el problema con los intrones artificiales es que si se agregan de manera indiscriminada, pueden provocar convergencia prematura, produciendo, en consecuencia, resultados muy pobres. Sin embargo, si su uso es debidamente controlado, pueden conducir a resultados excelentes, como parecen corroborar los experimentos de Angeline [16] El tercer problema suele ser aceptado sin mayor discusión, aún por los más fervientes defensores de los intrones: los intrones ocupan bastante memoria adicional y requieren un elevado tiempo de cómputo, lo que hace que el proceso evolutivo se torne más lento y que se consuman mayores recursos de cómputo de los debidos. Los efectos benéficos de los intrones Pero a pesar de sus efectos negativos, algunos investigadores argumentan que los intrones tienen cuando menos 2 efectos benéficos importantes, que bien pueden justificar su uso [1]
Tendencias futuras El trabajo actual sobre intrones gira en torno a las preguntas fundamentales que se plantean sobre ellos: ¿por qué se originan? y ¿cuál es su efecto en el proceso evolutivo? Si los intrones son una consecuencia natural de la programación genética, lo que nos debiera interesar entonces es minimizar sus efectos nocivos y tratar de aprovechar al máximo los positivos. Además, de ser benéficos para el proceso evolutivo, los intrones bien pudieran aprovecharse también en los algoritmos genéticos, aunque hasta ahora es bastante escaso el trabajo al respecto. Por lo que hemos visto en este artículo, los investigadores todavía no se ponen de acuerdo en torno a las respuestas a las incógnitas fundamentales que rodean a los intrones y, por ende, hay mucho campo libre para los investigadores interesados en abordar este tema. Se requieren más estudios teóricos y prácticos que validen las hipótesis existentes o que las desechen. Asimismo, se requieren aplicaciones más interesantes, en las que pueda demostrarse de manera convincente el efecto positivo de los intrones (si es que éste realmente existe), así como delinear con mayor precisión las situaciones en las cuales el uso de intrones será inevitablemente dañino para el proceso evolutivo. Aunque pocos investigadores lo mencionan, los estudios de los intrones en computación evolutiva, bien podrían traer beneficios (aunque fuesen accidentales) a la biología, en lo que podría convertirse en un curioso ejemplo de la naturaleza que imita al arte. Recursos en Internet Puede encontrarse información adicional sobre los intrones en computación evolutiva en las siguientes páginas: http://www.aic.nrl.navy.mil/~aswu: Página de Annie S. Wu, quien es una de las especialistas más connotadas en intrones y su papel en computación evolutiva (particularmente en los algoritmos genéticos). Annie ha publicado varios artículos sobre el tema (varios de los cuales están disponibles electrónicamente) y ha editado (junto con Wolfgang Banzhaf) un número especial dedicado a los intrones en la revista internacional "Evolutionary Computation" (publicada por el MIT Press), el cual se publicó en el invierno de 1998. http://ls11-www.informatik.uni-dortmund.de/people/banzhaf/: Página de Wolfgang Banzhaf, quien ha publicado varios artículos sobre los intrones y su papel en programación genética, además de ser co-autor de un libro introductorio a la programación genética [1], el cual incluye un capítulo sobre este tema. http://www.willamette.edu/~levenick: Página de James R. Levenick, quien fue el primero en abordar el tema de los intrones y su uso en los algoritmos genéticos. Su artículo de 1991 (considerado ahora como un clásico) está disponible electrónicamente desde esta página. [a] ADN significa ácido desoxirribonucleico [1] Banzhaf, Wolfgang, Nordin, Peter, Keller, Robert E. [8a] Levenick, James R. (1991) Inserting introns improves genetic algorithm success rate: Taking a clue from biology. In: R. K. Belew [2a] ] Wu, Annie S. [2b] Wu, Annie S. [b] los términos "intron" (intragenic region) y "exon" (expressed region) fueron acuñados por el biólogo Walter Gilbert en 1978 . [c] Según en Neo-darwinismo, toda la vida en el planeta puede ser explicada a través de un puñado de procesos estadísticos que actúan en y dentro de las poblaciones y especies: reproducción, mutación, competencia y selección. [d] Klyce hace una analogía entre los intrones y la aparición de comerciantes en una lengua extraña justo a la mitad de nuestro programa favorito de televisión. [5] Dawkins, Richard (1976) The Selfish Gene. Oxford University Press. [6] Blake, C. C. F. (1978) Do genes-in-pieces imply proteins-in-pieces? Nature, 273: p. 267. [3] Gilbert, Walter (1978) Why genes in pieces? Nature, February, 271(9): p. 501. [7] [7Gilbert, Walter, (1987) The Exon Theory of Genes, in Cold Spring Harbor Symposia on Quantitative Biology, Vol. LII: Evolution of Catalytic Function, pp. 907-913.] [8b] Levenick, James R. (1991) Inserting introns improves genetic algorithm success rate: Taking a clue from biology. In: R. K. Belew [e] Es importante hacer notar que el uso de intrones en las cadenas cromosómicas usadas en los algoritmos genéticos presupone el uso de un esquema de representación que permita el manejo de cadenas de longitud variable, y no de longitud fija como se acostumbra tradicionalmente en los algoritmos genéticos. [9] Wu, Annie S. [g] El término más usado en inglés para describir este fenómeno es "hitchhiking". La palabra "hitchhiking" se usa en inglés para denotar a las personas que piden "aventón" en las carreteras. En computación evolutiva , éste término se utiliza para denotar al fenómeno que hace que ciertos segmentos de cadena cromosómica que por sí mismos no tienen mayor relevancia, sean propagados debido a su similitud con los segmentos de cadena que tienen aptitudes elevadas. La correlación espuria existe también en la naturaleza y en computación evolutiva ha sido detectada desde principios de los 1990s. [10] Angeline, Peter J., (1994) Genetic Programming and Emergent Intelligence. In: K. E. Kinnear, Jr. (editor) Advances in Genetic Programming, The MIT Press. [11] Koza, John R. (1992) Genetic Programming. On the Programming of Computers by Means of Natural Selection, The MIT Press. [14] Tackett, W. A. (1994) Recombination, Selection, and the Genetic Construction of Computer Programs, PhD Thesis, University of Southern California, Department of Electrical Engineering Systems. [15] Nordin, Peter, Francone, Frank D. [1] Banzhaf, Wolfgang, Nordin, Peter, Keller, Robert E. [16] Angeline, Peter J. (1996) Two self-adaptive crossover operators for genetic programming. In: Angeline, Peter J. [1] Banzhaf, Wolfgang, Nordin, Peter, Keller, Robert E.
En este artículo hemos visto los conceptos básicos con respecto a los intrones biológicos y sus análogos en computación evolutiva. Además de definirlos y de proporcionar ejemplos de ellos en computación evolutiva, hemos tratado de compendiar, de manera breve, algunas de las respuestas que se han dado a las grandes interrogantes que rodean a los intrones en el contexto de la computación evolutiva. Asimismo, hemos discutido brevemente algunos de sus posibles efectos positivos y negativos y hemos planteado algunas de las rutas de investigación en esta área que lucen como más prometedoras, esperando así motivar a los interesados a explorar con más detenimiento este apasionante tema.
El primer autor agradece el apoyo del CONACyT a través del proyecto I-29870 A. Los tres autores restantes agradecen al CONACyT por haber recibido becas para cursar la Maestría en Inteligencia Artificial del LANIA y la Universidad Veracruzana.
, Banzhaf, Wolfgang, Nordin, Peter, Keller, Robert E. & Francone, Frank D. . (1998) Genetic Programming. An Introduction. Morgan Kaufmann Publishers.. , Wu, Annie S. & Lindsay, Robert K. (1996) . A Survey of Intron Research in Genetics, in Proceedings of the 4th Conference on Parallel Problem Solving from Nature, Berlin, Alemania, Springer-Verlag, pp. 101-110.. , Gilbert, Walter (1978) . Why genes in pieces? Nature, February, 271(9): p. 501.. Klyce, Brig, "Introns: A Mistery" [en línea]. http://www.panspermia.org/introns.htm. , Dawkins, Richard (1976) . The Selfish Gene. Oxford University Press.. , Blake, C. C. F. (1978) . Do genes-in-pieces imply proteins-in-pieces? Nature, 273: p. 267.. , Gilbert, Walter, (1987) . The Exon Theory of Genes, in Cold Spring Harbor Symposia on Quantitative Biology, Vol. LII: Evolution of Catalytic Function, pp. 907-913.. . , Levenick, James R. (1991) . Inserting introns improves genetic algorithm success rate: Taking a clue from biology. In: R. K. Belew & L. B. Booker (editors). Proceedings of the Fourth International Conference on Genetic Algorithms, pp. 123-127, Morgan Kaufmann.. , Wu, Annie S. & Lindsey, Robert K. (1995) . Empirical studies of the genetic algorithm with noncoding segments. Evolutionary Computation, 3(2): 121-147.. , Angeline, Peter J., (1994) Genetic Programming and Emergent Intelligence. In: K. E. Kinnear, Jr. (editor) Advances in Genetic Programming, The MIT Press. . Genetic Programming and Emergent Intelligence. In: K. E. Kinnear, Jr. (editor) Advances in Genetic Programming, The MIT Press.. , Koza, John R. (1992) . Genetic Programming. On the Programming of Computers by Means of Natural Selection, The MIT Press.. , Das, Rajarshi & Whitley, Darrell (1991) . The Only Challenging Problems are Deceptive: Global Search by Solving Order-1 Hyperplanes. In: Richard K. Belew & Lashoon B. Booker (editors) Proceedings of the Fourth International Conference on Genetic Algorithms, pp. 166-173, Morgan Kaufmann Publishers.. , Schaffer, J. David, Eshelman, Larry J. & Offutt, Daniel (1991) . Spurious Correlations and Premature Convergence in Genetic Algorithms". In: Gregory J. E. Rawlins (editor) Foundations of Genetic Algorithms, pp. 102-112, Morgan Kaufmann Publishers.. , Tackett, W. A. (1994) . Recombination, Selection, and the Genetic Construction of Computer Programs, PhD Thesis, University of Southern California, Department of Electrical Engineering Systems.. , Nordin, Peter, Francone, Frank D. & Banzhaf, Wolfgang (1995) . Explicitly defined introns and destructive crossover in genetic programming. In: Justinian P. Rosca (editor) Proceedings of the Workshop on Genetic Programming: From Theory to Real-World Applications, Tahoe City, California, pp. 6-22..
, Angeline, Peter J. (1996) . Two self-adaptive crossover operators for genetic programming. In: Angeline, Peter J. & Kinnear, Jr. Kenneth E. (editors) Advances in Genetic Programming 2, Chapter 5, pp. 89-110, MIT Press.. , Andre, David & Teller, Astro (1996) A study in program response and the negative effects of introns in genetic programming. In: Koza, John R., Goldberg, David E., Fogel, David B. & Riolo, Rick L. (editors) Genetic Programming 1996: Proceedings of the First Annual Conference, pp. 12-20, MIT Press. . A study in program response and the negative effects of introns in genetic programming. In: Koza, John R., Goldberg, David E., Fogel, David B. & Riolo, Rick L. (editors) Genetic Programming 1996: Proceedings of the First Annual Conference, pp. 12-20, MIT Press.. |
|
[ Este número ] |
|
Dirección General de Servicios de Cómputo
Académico-UNAM |