

Revista Digital Universitaria ISSN: 1607 - 6079 | Publicación mensual
1 de febrero de 2015 vol.16, No.2
• TEMA DEL MES •
Cultura, patrimonio y futuro del frijol en México
Introducción
Dice la Madre Tierra a Watákame al prepararlo para el diluvio: “Guarda contigo cinco granos de maíz de cada color y cinco semillas de frijol, también de cada color”
Alfredo López Austin, Los mitos del tlacuache: caminos de la mitología mesoamericana
México es reconocido como centro primario de domesticación y diversidad genética del frijol (GEPTS y DEBOUCK, 1991). En el país se conoce como frijol a diferentes especies del género Phaseolus, entre las cuales, las de mayor importancia económica son: P. vulgaris (frijol común), P. coccineus (frijol ayocote), P. lunatus (frijol lima) y P. acutifolius (frijol tépari).
Desde que fue domesticado, el frijol combinado con el maíz ha constituido la base de la alimentación de los mexicanos. Esta leguminosa se ha cultivado en América durante miles de años (BRICK y BURGENER, 2007), debido a su disponibilidad y al hecho de que es una excelente fuente de proteínas, hidratos de carbono, fibra y minerales. En algunos países, como México y Brasil, y los que rodean los Grandes Lagos en África Oriental (Burundi, Rwanda, Kenya, Tanzania), los frijoles son la principal fuente de proteína en la dieta humana, ya que contienen aminoácidos esenciales como lisina, treonina, valina, isoleucina y leucina (MA y BLISS, 1978), y esto los sitúa entre las especies ideales para estudios de nutrición (FAO: http://faostat.fao.org/). Existen numerosos estudios clínicos que avalan que el consumo regular de frijol ayuda a la prevención y tratamiento de enfermedades cardiovasculares, diabetes mellitus, obesidad y enfermedades relacionadas con el aparato digestivo, ya que reduce el nivel de colesterol y glucosa en la sangre (SINGH, 1992; THOMPSON et al., 2009; BENNINK, 2010).
Además de su calidad como alimento, el frijol desempeña una importante tarea, ya que posee la capacidad de fijar nitrógeno en el suelo, dado que forma asociaciones simbióticas con bacterias que fijan nitrógeno atmosférico. De ahí su importancia en la milpa, agroecosistema mesoamericano originario de la tradición maya, cuyos principales componentes productivos son maíz, frijol y calabaza, denominados "las tres hermanas" por la manera en que se complementan. La caña del maíz sirve como soporte al frijol trepador. A su vez, el frijol aporta nitrógeno en la parte subterránea, elemento necesario para el crecimiento del maíz. Las grandes hojas de la calabaza cubren la parte superficial del suelo y ayudan a mantenerlo libre de plantas no deseadas, contribuyendo además a disminuir la evaporación del agua en la superficie. A pesar de que muchas y diversas asociaciones contribuyen a la fijación simbiótica del nitrógeno, en la mayor parte de los sistemas agrícolas la fuente primaria (80%) de nitrógeno fijado se debe a la simbiosis Rhizobium-leguminosa.
El frijol es fundamental en la gastronomía y en la cultura mexicana y, sin embargo, su cultivo ha sido desplazado en muchos casos a regiones marginales, lo que ha derivado en pérdidas de diversidad genética, reducción de rendimiento y disminución de competitividad de este cultivo (BELLÓN et al., 2009). La diversidad genética existente en las poblaciones silvestres del frijol, así como en otros parientes silvestres del género Phaseolus, constituye un recurso importante para el fitomejoramiento, ya que dichas poblaciones albergan genes de tolerancia a factores ambientales adversos (frío, sequía, calor, enfermedades y plagas), que podrían ser empleados para ampliar la base genética del frijol cultivado en programas de mejora.
El Banco de Germoplasma de Frijol del Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP), ubicado en el Estado de México, alberga más de 7,000 registros de frijoles cultivados (VARGAS et al., 2006), sin embargo, las formas silvestres están poco representadas. Esta situación se repite en otros bancos de germoplasma mundiales. Según la información recogida en el portal Genesys (http://www.genesys-pgr.org), existen unas 83,000 entradas almacenadas en bancos de germoplasma de diferentes países, de las cuales sólo el 2% son silvestres.
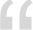 |
El frijol es fundamental en la gastronomía y en la cultura mexicana y, sin embargo, su cultivo ha sido desplazado en muchos casos a regiones marginales... | |
 |
||
La mejora asistida por marcadores moleculares (MAS, del inglés Marker-Assisted Selection) es una importante herramienta en los programas de mejoramiento genético de plantas, debido a que aumenta la eficiencia y efectividad en comparación con los métodos tradicionales. Una vez que se identifican los marcadores específicos de ADN que están estrechamente ligados a un gen o QTL (del inglés Quantitative Trait Loci; Loci de carácter cuantitativo) responsables de una característica de interés para la mejora, dichos marcadores se pueden usar para identificar aquellas plantas que albergan esos genes o QTLs, y evitar así una larga y laboriosa evaluación de campo. Además, muchos caracteres, como el rendimiento, valor nutritivo o resistencia a diferentes estreses, son muy complejos y, por ello, difíciles de evaluar en el campo.
Los avances en la secuenciación genómica aportan la oportunidad de desarrollar millones de marcadores e identificar genes de importancia agronómica, y permiten, por tanto, conocer cómo se comportan los caracteres. Dichos marcadores pueden usarse en programas de mejora, análisis de diversidad genética, identificación de cultivares, análisis filogenéticos, caracterización de recursos genéticos y asociación con caracteres agronómicos (EDWARS y BATLEY, 2010). La disponibilidad de un genoma de referencia de la especie en cuestión, permite la rápida identificación de genes candidatos a través de análisis bioinformático y la detección de marcadores tipo SNP (del inglés Single Nucleotide Polymorphism, polimorfismo de un sólo nucleótido) a través de la comparación de dicho genoma con secuencias de diferentes genotipos de la especie.
Por lo tanto, es indispensable la conservación de los parientes silvestres y de las variedades tradicionales de frijol, que podrán ser empleadas en programas de mejoramiento genético de la especie, a través de herramientas genómicas, las cuales permitirán un máximo aprovechamiento de la riqueza genética para asegurar diversidad suficiente y poder afrontar así las futuras demandas de la producción agrícola ante condiciones climáticas impredecibles.
Historia y origen del frijol
El frijol común (Phaseolus vulgaris L.) es una especie autógama, diploide y con un genoma relativamente pequeño, con un tamaño estimado entre 587 Mbp y 637 Mbp (ARUMUGANATHAN y EARLE, 1991; BENNETT y LEITCH, 1995, 2010).De acuerdo con la distribución geográfica de la mayoría de especies (ver Figura 1) que pertenecen al género Phaseolus, éstas son consideradas de origen mesoamericano (FREYTAG y DEBOUCK 1996, 2002; DELGADO-SALINAS et al., 1999, 2006). Delgado-Salinas et al. (1999) clasificaron el género Phaseolus en 50 especies, mientras que Freytag y Debouck (2002) en 70 especies. De todas ellas, sólo cinco han sido domesticadas: P. acutifolius A. Gray, P. coccineus L., P. lunatus L., P. dumosus Macfad. y P. vulgaris L., siendo esta última la de mayor importancia económica.
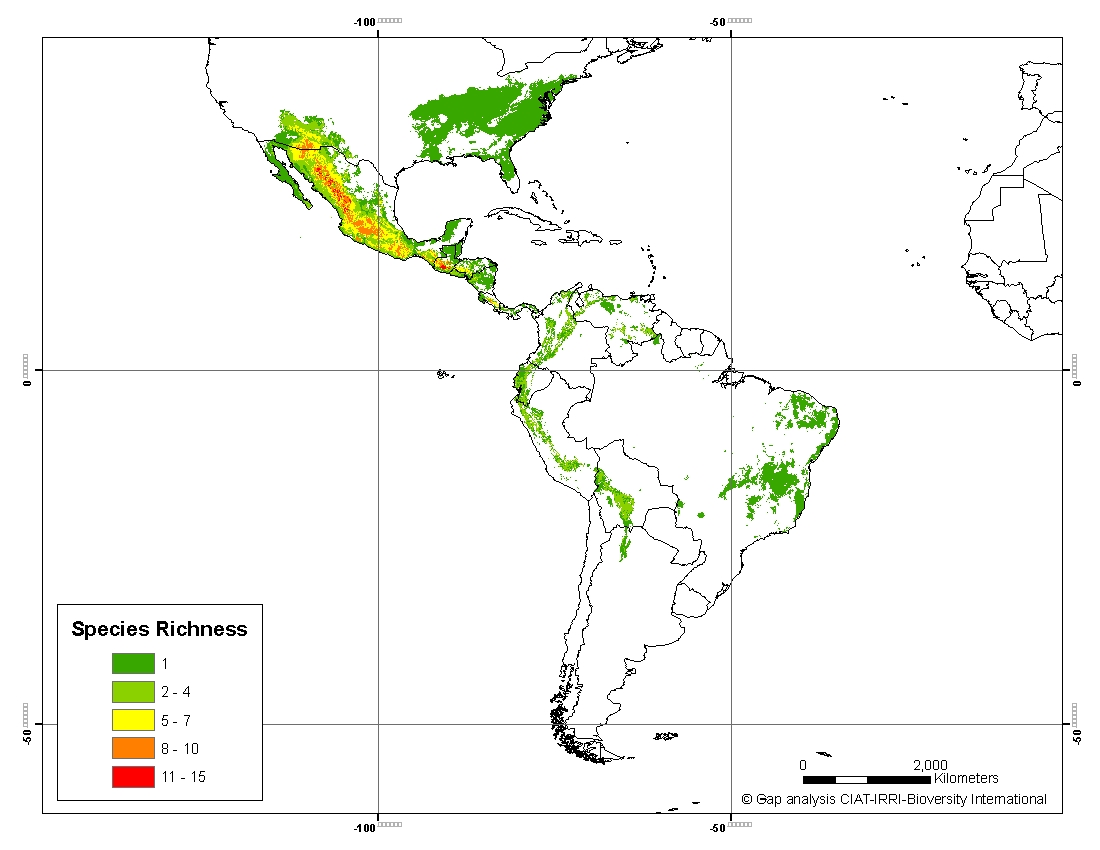
Figura 1. Distribución de la riqueza de especies del género Phaseolus en el continente americano. La leyenda indica el número de especies asociado a cada color presente en el mapa. Fuente: http://gisweb.ciat.cgiar.org/GapAnalysis/
Las formas silvestres de frijol están ampliamente distribuidas desde Chihuahua hasta San Luis, en Argentina (TORO et al., 1990), y pueden clasificarse en dos acervos genéticos: Mesoamericano (del Norte de México a Colombia) y Andino (Sur de Perú, Bolivia y Noroeste de Argentina), de acuerdo con su origen geográfico, la morfología de la planta (SINGH et al., 1991), proteínas de la semilla (GEPTS et al., 1986; GEPTS y BLISS, 1985), alozimas (KOENIG y GEPTS, 1989) y marcadores moleculares (BECERRA-VELÁSQUEZ y GEPTS, 1994; FREYRE et al., 1996; PAPA y GEPTS 2003, ROSSI et al., 2009, KWAK y GEPTS, 2009). Según los restos arqueológicos, la domesticación y el primer consumo sucedió hace 6,000-8,000 años en Mesoamérica y Sudamérica (BRICK y BURGENER, 2007).
Debouck et al. (1993) descubrieron poblaciones silvestres de P. vulgaris en el norte de Perú y Ecuador en la década de los ochenta. Más tarde, Kami et al. (1995) identificaron una nueva faseolina (proteína de reserva de la semilla) en dichas poblaciones, que no se encontraba en las pertenecientes a los acervos genéticos Mesoamericano y Andino, y sugirieron que esta faseolina era ancestral a las otras ya conocidas, por lo que se estableció la hipótesis de que el origen del frijol común estaba en la zona del Norte de Perú y Ecuador, desde donde las formas silvestres se dispersaron posteriormente hacia el norte (Colombia, América Central y México) y al sur (Sur de Perú, Bolivia y Argentina), formando los dos acervos genéticos conocidos en la actualidad. Sin embargo, esta hipótesis ha sido debatida por múltiples estudios, en particular el llevado a cabo por Bitocchi et al. (2012), el cual, mediante el estudio de la diversidad de nucleótidos en cinco fragmentos génicos, indicó un claro origen Mesoamericano de P. vulgaris, principalmente mexicano.
Domesticación del frijol
La domesticación a partir de especies silvestres se inició en diferentes partes del mundo hace unos 12,000 años, convirtiéndose en uno de los hechos más importantes de la historia de la humanidad. El cultivo provocó una selección artificial de las plantas que llevó al desarrollo de cultivos modernos, adaptados a ambientes artificiales. Durante la domesticación, el frijol ha sufrido una multitud de cambios tanto morfológicos como fisiológicos que lo diferencian de sus parientes silvestres, como la dormancia de la semilla, la dehiscencia del fruto, el hábito de crecimiento, la sensibilidad al fotoperiodo y color, y la forma y talla tanto de la planta como de las semillas y frutos (Figura 2). Todos estos caracteres son denominados colectivamente como síndrome de domesticación (HAMMER, 1984). Una de las consecuencias de la domesticación en la mayoría de las especies de cultivo, es la reducción de la diversidad genética (GLÉMIN y BATAILLON, 2009).
Figura 2. Diferencias de vainas y semillas entre genotipos domesticados y silvestres de P. vulgaris. a y b, domesticados; c, silvestre Andino; d y e, silvestres Mesoamericanos. Se observan las diferencias en tamaño, tanto de vaina como de semilla y la torsión de las vainas en los genotipos silvestres, que presentan dehiscencia explosiva. Barra blanca: 1 cm.
A pesar de que las poblaciones silvestres del acervo Mesoamericano muestran una elevada diversidad genética en comparación con las Andinas, diversos estudios han analizado la pérdida de diversidad debida a la domesticación (KOENIG y GEPTS, 1989; FREYRE et al., 1996.; ROSSI et al., 2009; KWAK y GEPTS, 2009; NANNI et al., 2011; BITOCCHI et al., 2012, 2013; DESIDERIO et al., 2013), concluyendo que, cuando se comparan poblaciones silvestres frente a domesticadas, la pérdida de diversidad es mayor en Mesoamérica que en los Andes. Según lo propuesto por Bitocchi et al., (2013), esta diferencia puede explicarse como el resultado de los cuellos de botella que se produjeron antes de la domesticación en los Andes (ROSSI et al., 2009; BITOCCHI et al., 2012), lo que empobreció fuertemente las poblaciones silvestres, dando lugar a los efectos secundarios del cuello de botella posterior a la domesticación.
Otro estudio llevado a cabo por Bellucci et al. (2014) compara datos transcriptómicos de frijoles silvestres y domesticados, y observa una reducción de la diversidad en las formas domesticadas con respecto a las silvestres. Sin embargo, también señala que la diversidad en las variedades tradicionales o criollas que se originó por la fijación de mutaciones útiles después de la domesticación es una fuente importante para la mejora de los cultivos.
Todo esto indica la importancia del estudio de la evolución de una especie de cultivo como un factor que influye en la diversidad genética actual. Además, señala que la gestión eficiente de los recursos genéticos disponibles depende de la posesión de información genética detallada de la especie, la cual se vio afectada por el proceso de domesticación, así como la necesidad de conservar las poblaciones silvestres y criollas de frijol como fuente de mejora para el cultivo (KELLY et al., 1998; GEPTS, 2004).
Importancia económica
El frijol común se considera la leguminosa de grano destinada al consumo humano directo más importante del mundo (MIKLAS y SINGH, 2007; BROUGHTON et al., 2003). Los seis principales países productores en el mundo son: Brasil, India, China, Myanmar, México y Estados Unidos de América, en ese orden. Desde 1999, Brasil es el primer país productor en el mundo (http://apps.fao.org/faostat ). Según datos de la FAO, México ocupaba en el año 2012 el sexto lugar, con India y Brasil a la cabeza.
Frijol común. Imagen de: Victor Flores. Por su gran importancia económica y social, el frijol es un producto estratégico dentro del desarrollo rural de México. Los 10 principales estados productores son: Zacatecas, Sinaloa, Chihuahua, Durango, Nayarit, Chiapas, Guanajuato, San Luis Potosí, Puebla e Hidalgo.
En México, la superficie anual sembrada con frijol es de 1.5 millones de hectáreas (Instituto Nacional de Estadística y Geografía, 2011), con una producción promedio de 0.7 toneladas por hectárea –en contraste con las 1.86 ton/ha producidas en los Estados Unidos–, y como promedio, cada mexicano consume anualmente 10.30 kg (FAO), por lo que este cultivo se considera el segundo en importancia en México, con base en la superficie sembrada y el volumen consumido. Además, representa la segunda actividad agrícola más relevante en el país por el número de productores dedicados al cultivo.
Mejora genética del frijol
Debido a su importancia como alimento, el valor del frijol común se contempla como un cultivo social y su mejora es una preocupación constante (SINGH, 2001).Se considera que la mejora genética vegetal comenzó con la domesticación de los primeros cultivos. Inicialmente, los programas de mejora genética en frijol empleaban métodos tradicionales o convencionales, basados fundamentalmente en la genética mendeliana. Estos consisten en el cruzamiento entre individuos de la misma especie (o entre especies afines) que muestran características diferentes, y una selección posterior de los ejemplares que presentan las características deseadas. Dicho proceso se repite sucesivamente hasta lograr la incorporación de los genes que llevan información para los rasgos ambicionados y la eliminación de aquellos relacionados con las características no deseadas. Más recientemente, estos programas se han visto beneficiados por la aplicación de técnicas de genética molecular, como es la mejora asistida por marcadores (MAS), la cual requiere que previamente se haya realizado la identificación, etiquetado y mapeo de loci de QTLs de interés (MIKLAS et al., 2006). De forma general, las ventajas de la MAS incluyen una reducción en el tiempo y esfuerzo, y una mayor eficiencia (DWIVEDI et al., 2007; JENA y MACKILL, 2008).
La mayoría de estudios de MAS en frijol común se ha enfocado en caracteres de resistencia a diversas enfermedades bacterianas, víricas y fúngicas (KELLY et al., 2003). Para el empleo de la MAS se requiere la existencia de un mapa de ligamiento genético, es decir, representaciones gráficas que muestren el orden de los genes en un cromosoma y donde dicho orden esté basado en las frecuencias de recombinación entre los genes. Se ha desarrollado una multitud de mapas de ligamiento de frijol, inter y entre acervos genéticos. Hasta la fecha hay unos 25 mapas publicados (BELLUCCI et al., 2014), entre ellos el de la población BAT93/Jalo EPP558, considerada como la población de mapeo consenso de frijol ya que en ella se han mapeado multitudes de genes y QTLs de importancia agronómica de otros mapas, permitiendo entonces la correlación entre grupos de ligamiento (FREYRE et al., 1998; VALLEJOS et al., 2001; BLAIR et al., 2003). Los mapas de ligamiento se emplean en una gran cantidad de propuestas: mapeo de genes, análisis de QTLs, análisis de desequilibrio de ligamiento, sintenia y, por tanto, para encontrar genes relacionados con caracteres de interés agronómicos, para su posterior aplicación en programas de mejoramiento. Algunos ejemplos en los que la MAS se ha empleado con éxito en programas de mejoramiento de diferentes especies de cultivo se resumen en la Tabla 1.
Tabla 1. Cultivares desarrollados mediante MAS disponibles en el mercado. Casos exitosos llevados a cabo por instituciones públicas.
Genómica: herramienta para la mejora
Se ha demostrado que la mejora de los cultivos se puede agilizar mediante el aprovechamiento de las tecnologías de genómica de alto rendimiento que están teniendo efectos significativos en la gestión de los bancos de genes y en la manera de explotar los recursos de germoplasma (TUBEROSA et al., 2011). |
Futuros estudios de genómica facilitarán la obtención de conocimientos más precisos sobre el origen, domesticación, evolución, adaptación y mejora de la especie... | |
 |
||
La MAS es utilizada de forma rutinaria para los rasgos controlados por los principales loci, aunque la mejora de caracteres cuantitativos complejos todavía sigue siendo una tarea difícil en los programas de mejoramiento (MIKLAS et al., 2006a; TUBEROSA et al., 2011). El estudio de asociación del genoma completo (GWAS, Genome-wide association study) es un enfoque que está siendo adoptado cada vez más para diseccionar la base genética de los caracteres, y cuando se aplica a las poblaciones silvestres, tiene importantes beneficios para la conservación genética (ALLENDORF et al., 2010; GALEANO et al., 2012). Además, las nuevas técnicas de la genómica pueden ser empleadas para estudios de domesticación, adaptación de las especies, deriva genética y flujo de genes (DAVEY et al., 2011; HEERWAARDEN et al., 2011).
Conclusiones
Hasta la fecha, la falta de un genoma de referencia del frijol común ha sido uno de los principales limitantes para los programas de mejora. La disponibilidad del genoma de referencia del cultivo permitirá perfeccionar la competitividad de esta especie, ya que la secuencia proporcionará herramientas de gran alcance para mejorar características agronómicas y nutricionales. Futuros estudios de genómica facilitarán la obtención de conocimientos más precisos sobre el origen, domesticación, evolución, adaptación y mejora de la especie, así como de las estrategias para su conservación.Por otro lado, es inminente la necesidad de generar un Banco de Germoplasma bien caracterizado y accesible de la especie en México, teniendo en cuenta la conservación de material silvestre, criollo y domesticado, tanto in situ como ex situ, y preservar así el valor social de este cultivo.
Bibliografía
ALLENDORF, F.W., P. A. Hohenlohe y G. Luikart. “Genomics and the future of conservation genetics”, Nat Rev Genet, 2010, 11, pp. 697-709.
ARUMUGANTHAN, K., E. Earle. “Nuclear DNA content of some important plant species”, Plant Mol Biol Rep, 1991, 9, pp. 208-218.
BECERRA-VELÁSQUEZ, V.L. y P. Gepts. “RFLP diversity in common bean (Phaseolus vulgaris L.)”, Genome, 1994, 37, pp. 256-263.
BELLÓN, M.R. et al. “Diversidad y conservación de recursos genéticos en plantas cultivadas”, en Capital natural de México, vol. II: Estado de conservación y tendencias de cambio. México: CONABIO, 2009, pp. 355-382.
BELLUCCI, E. et al. “Genomics of Origin, Domestication and Evolution of Phaseolus vulgaris”, R. Tuberosa et al. (Eds.), Genomics of Plant Genetic Resources, Springer Science+Business Media Dordrecht, 2014, Chapter 20.
BELLUCCI, E. et al. “Decreased Nucleotide and Expression Diversity and Modified Coexpression Patterns Characterize Domestication in the Common Bean”, Plant Cell, 2014, doi: 10.1105/tpc.114.124040.
BENNETT, M.D. y I.J. Leitch. “Nuclear DNA amounts in angiosperms”, Ann Bot, 1995, 76, pp. 113-116.
“Angiosperm DNA C-values database” (release 8.0, December 2012) http://data.kew.org/cvalues/
BENNINK, M.R. “Health benefits associated with consumption of dry beans”, Annu Rep Bean Improv Coop, 2010, 53, pp. 2-3.
BITOCCHI, E. et al. “Mesoamerican origin of the common bean (Phaseolus vulgaris L.) is revealed by sequence data”, Proc Natl Acad Sci USA, 2012, 109(14), pp. E788–E796.
BITOCCHI, E. et al. “Molecular analysis of the parallel domestication of the common bean in Mesoamerica and the Andes”, New Phytol, 2013, 197, pp. 300-313.
BLAIR, M.W. et al. “Development of a genome-wide anchored microsatellite map for common bean (Phaseolus vulgaris L.)”, Theor Appl Genet, 2003, 107, pp. 1362-1374.
BRICK, M.A. y P. Burgener. “Colorado Beans News. Origin and World Production”, 2007, Vol. 20(1), pp.7 [en línea]: www.csuag.com/cbn
BROUGHTON, W.J. et al. “Beans (Phaseolus spp.)—model food legumes”, Plant Soil, 2003, 252, pp. 55-128.
CHENG, S.H. et al. “Progress in research and development on hybrid rice: a super-domesticate in China”, Ann Bot, 2007, 100, pp. 959-966.
DAVEY, J.W. et al. “Genome–wide genetic marker discovery and genotyping using next–generation sequencing”, Nat Rev Genet, 2011, 12, pp. 499-510.
DEBOUCK, D.G. et al. “Genetic diversity and ecological distribution of Phaseolus vulgaris in northwestern South America”, Econ Bot, 1993, 47, pp. 408-423.
DELGADO-SALINAS, A., R. Bibler y M. Lavin. “Phylogeny of the genus Phaseolus (Leguminosae): a recent diversification in an ancient landscape”, Syst Bot, 2006, 31, pp. 779-791.
DELGADO-SALINAS, A., T. Turley, A. Richman y M. Lavin. “Phylogenetic analysis of the cultivated and wild species of Phaseolus (Fabaceae)”, Syst Bot, 1999, 24(3), pp. 438-460.
DESIDERIO, F. et al. “Chloroplast microsatellite diversity in Phaseolus vulgaris”, Front Plant Sci, 2013, 3, pp. 312.
DWIVEDI, S.L. et al. “The molecularization of public sector crop breeding: Progress, problems, and prospects”, Adv Agron, 2007, 95, pp. 163-318.
EDWARDS, D. y J. Batley. “Plant genome sequencing: applications for crop improvement”, Plant Biotechnol J, 2010, 8(1), pp. 2-9.
FAO, Food and Agriculture Organization of the United Nations. “Food Balance Sheet 2012” [en línea]: http://faostat.fao.org [Consulta: 20 abril 2014].
FREYRE, R. et al. “Ecogeographic distribution of Phaseolus spp. (Fabaceae) in Bolivia”, Econ Bot, 1996, 50, pp. 195-215.
FREYRE, R. et al. “Towards an integrated linkage map of common bean. 4. Development of a core map and alignment of RFLP maps”, Theor Appl Genet, 1998, 97, pp. 847-856.
FREYTAG, G.F. y D.G. Debouck. “Phaseolus costaricensis, a new wild bean species (Phaseolinae, Leguminosae) from Costa Rica and Panama, central America”, Novon, 1996, 6, pp. 157-163.
FREYTAG, G.F. y D.G. Debouck. Taxonomy, distribution, and ecology of the genus Phaseolus (Leguminosae–Papilionoideae) in North America, Mexico and Central America. Texas: Botanical Research Institute of Texas, Ft. Worth. BRIT, 2002.
GALEANO, C. et al. “Gene–based single nucleotide polymorphism markers for genetic and association mapping in common bean”, BMC Genet, 2012, 13(1), pp. 48.
GANDHI, D. “UAS scientist develops first drought tolerant rice”, [en línea] The Hindu. 17 noviembre 2007. http://www.thehindu.com/todays-paper/tp-national/tp-karnataka/uas-scientist-develops-first-drought-tolerant-rice/article1949838.ece. [Consulta: 20 abril 2014].
GENESYS. “Gateway to Plant Genetic Resources”, http://www.genesys-pgr.org [Consulta: 20 abril 2014].
GEPTS, P. “Domestication as a long‐term selection experiment”, Plant Breed Rev, 2004, 24 (Parte 2), pp. 1‐44.
GEPTS, P. et al. “Phaseolin–protein variability in wild forms and landraces of the common bean (Phaseolus vulgaris): evidence for multiple centers of domestication”, Econ Bot, 1986, 40, pp. 451-468.
GEPTS, P. et al. “Genomics of Phaseolus beans, a major source of dietary protein and micronutrients in the Tropics”, En: P.H. Moore y R. Ming (Eds.), Genomics of Tropical Crop Plants, Berlin: Springer, 2008, pp. 113-143.
GEPTS, P. y F.A. Bliss. “F1 hybrid weakness in the common bean: differential geographic origin suggests two gene pools in cultivated bean germplasm”, J Hered, 1985, 76, pp. 447-450.
GEPTS, P. y D.G. Debouck. “Origin, domestication, and evolution of the common bean (Phaseolus vulgaris L.)”, En: A.V. Schoonhoven y O. Voysest (Eds.) Common beans: Research for crop improvement, Wallingford, Oxon, UK: CIAT-C.A.B. International, 1991, pp. 7-53.
GLÉMIN, S. y T. Bataillon. “A comparative view of the evolution of grasses under domestication”, New Phytol, 2009, 183, pp. 273-290.
GUPTA, H.S. et al. “Quality protein maize for nutritional security: rapid development of short duration hybrids through molecular marker assisted breeding”, Curr Sci, 2009, 96(2), pp. 230-236.
HAMMER, K. “Das Domestikationssyndrom”, Kulturpflanze, 32, 1984, pp. 11-34.
HEERWAARDEN, J. Van et al. “Genetic signals of origin, spread, and introgression in a large sample of maize landraces”, Proc Natl Acad Sci USA, 2011, 108, pp. 1088-1092.
INEGI, Instituto Nacional de Estadística y Geografía [en línea]: http://www.inegi.org.mx [Consulta: 20 abril 2014].
JENA, K.K. y D.J. Mackill. “Molecular markers and their use in marker-assisted selection in rice”, Crop Sci, 2008, 48, pp. 1266-1267.
KAMI, J. et al. “Identification of presumed ancestral DNA sequences of phaseolin in Phaseolus vulgaris”, Proc Natl Acad Sci USA, 1995, 92, pp. 1101-1104.
KELLY, J.D. et al. “Tagging and mapping of genes and QTL and molecular–marker assisted selection for traits of economic importance in bean and cowpea”, Field Crops Res, 2003, 82, pp. 135-154.
KELLY, J.D., J.M. Kolkman y K. Schneider. “Breeding for yield in dry bean (Phaseolus vulgaris L.)”, Euphytica, 1998, 102, pp. 343‐356.
KOENIG, R. y P. Gepts. “Allozyme diversity in wild Phaseolus vulgaris: further evidence for two major centers of diversity”, Theor Appl Genet, 1989, 78, pp. 809-817.
KWAK, M. y P. Gepts. “Structure of genetic diversity in the two major gene pools of common bean (Phaseolus vulgaris L., Fabaceae)”, Theor Appl Genet, 2009, 118, pp. 979-992.
LIPPMAN, Z.B., Y. Semel y D. Zamir. “An integrated view of quantitative trait variation using tomato interspecific introgression lines”, Curr Opin Genet Dev, 2007, 6, pp. 545-552.
MA, Y. y F.A. BLISS. “Seed Proteins of Common Bean”, Crop Sci<, 1978, 18, pp. 431-437.
McCLEAN, P.E. et al. “Phaseolus vulgaris: a diploid model for soybean”, En: Stacey, G. (Eds.) Soybean Genomics, Berlin: Springer, 2008, pp. 55-78.
MIKLAS, P.N., J.D. Kelly y S.P. Singh. “Registration of anthracnose-resistant pinto bean germplasm line USPT-ANT-1”, Crop Sci, 2003, 43, pp. 1889-1890.
MIKLAS, P.N., J.D. Kelly, S.E. Beebe y M.W. Blair. “Common bean breeding for resistance against biotic and abiotic stresses: from classical to MAS breeding”, Euphytica, 2006a, 147, pp. 105-131.
MIKLAS, P.N., J.R. Smith y S.P. Singh. “Registration of common bacterial blight resistant dark red kidney bean germplasm line USDK-CBB-15”, Crop Sci, 2006b, 46, pp. 1005-1006.
MIKLAS, P.N. y S.P. Singh. “Common Bean. Genome Mapping and Molecular Breeding in Plants”, En: Kole, Chittaranjan (Eds.) Pulses, Sugar and Tuber Crops, Berlin, Heidelberg: Springer-Verlag, 2007, 3, pp. 1-31.
MUTLU, N. et al. “Registration of pinto bean germplasm line ABCP-8 with resistance to common bacterial blight”, Crop Sci, 2005, 45, pp. 806.
MUTLU, N. et al. “Registration of common bacterial blight, rust and bean common mosaic resistant great northern bean germplasm line ABC – Weighing”. J Plant Regist, 2008, 2, pp. 120-124.
NANNI, L. et al. “Nucleotide diversity of a genomic sequence similar to SHATTERPROOF (PvSHP1) in domesticated and wild common bean (Phaseolus vulgaris L.)”, Theor Appl Genet, 2011, 123, pp. 1341-1357.
PAPA, R. y P. Gepts. “Asymmetry of gene flow and differential geographical structure of molecular diversity in wild and domesticated common bean (Phaseolus vulgaris L.) from Mesoamerica”, Theor Appl Genet, 2003, 106, pp. 239-250.
ROSSI, M. et al. “Linkage disequilibrium and population structure in wild and domesticated populations of Phaseolus vulgaris L.” Evol Appl, 2009, 2, pp. 504-522.
SCHMUTZ, J. et al. “A reference genome for common bean and genome-wide analysis of dual domestications”, Nat Genet, 2014, doi:10.1038/ng.3008.
SINGH, S.P. “Common bean improvement in the tropics”, Plant Breed Rev, 1992, 10, pp. 199-269.
SINGH, S.P. “Broadening the genetic base of common bean cultivars”, Crop Sci, 2001, 41, pp. 1659-1675.
SINGH, S.P. et al. “Genetic diversity in cultivated common bean. II. Marker–based analysis of morphological and agronomic traits”. Crop Sci, 31, (1991 pp. 23-29.
THOMPSON, M.D., M.A. Brick, J.N. McGinley y H.J. Thompson. “Chemical composition and mammary cancer inhibitory activity of dry beans”. Crop Sci, 49, (2009 pp. 179-186.
TORO, O., J. Tohme y D.G. Debouck. Wild bean (Phaseolus vulgaris L.): Description and distribution. Cali, Colombia: International Board of Plant Genetic Resources (IBPGR) and Centro Internacional de Agricultura Tropical (CIAT), 1990, pp. 106.
TUBEROSA, R., A. Graner y R.K. Varshney. “Genomics of plant genetic resources: an introduction”, Plant Genet Resour, 2011, 9, pp. 151-154.
VALLEJOS, C., P. Skroch y J. Nienhuis. “Phaseolus vulgaris – The common bean. Integration of RFLP and RAPD-based linkage maps”. En: R. Phillips y I. Vasil (Eds.), DNA-based markers in plants, The Netherlands: Kluwer, Dordrecht, , 2001, pp. 301-317.
VARGAS, M.L.P. et al. “Colección Núcleo de Phaseolus vulgaris L. del INIFAP. Catálogo de Accesiones de la Forma Domesticada”. Libro Técnico Núm. 10, Chapingo, México: CEVAMEX–SAGARPA, 2006, pp. 461.
WheatCAP, Wheat Coordinated Agricultural Project. Germplasm release 2007. University of California, Davis. http://maswheat.ucdavis.edu/Achievements/cultivars2007.htm [Consulta: 22 abril 2014].
COMENTARIOS
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO
Dirección General de Cómputo y de Tecnologías de Información y Comunicación
Dirección General de Cómputo y de Tecnologías de Información y Comunicación
2013 Esta obra está bajo una licencia de Creative Commons 















