CRISPR-Cas: la nueva herramienta para diagnosticar enfermedades infecciosas
Vol. 22, núm. 5 septiembre-octubre 2021
CRISPR-Cas: la nueva herramienta para diagnosticar enfermedades infecciosas
Jorge Melendez Zajgla, Bertha Rueda-Zarazúa, Alfredo Garcia Venzor y Vilma Maldonado CitaResumen
En este artículo se presenta un nuevo uso para la tecnología de edición genómica crispr-Cas, cuyo desarrollo le permitió ganar el premio Nobel de Química 2020 a las doctoras Jennifer Doudna y Emmanuelle Charpentier. Esta herramienta no sólo permite modificar secuencias de adn, sino que también se puede utilizar como una prueba muy rápida y sensible para el diagnóstico clínico. Debido a la reciente pandemia, se ha generado un crecimiento exponencial de métodos para detectar sars-CoV-2, causante de la covid-19, entre los que destaca el uso de crispr-Cas. Esta tecnología nos permitió crear un ensayo rápido y económico que puede ser implementado fácilmente para aumentar el número de pruebas realizadas y así contribuir con un mejor control de la pandemia. Su papel en el diagnóstico molecular presenta gran potencial no sólo para detectar la presencia de virus y bacterias, sino incluso para diagnosticar enfermedades no infecciosas como el cáncer.
Palabras clave: edición genética, diagnóstico, detección, covid-19, sars-CoV-2.
CRISPR-Cas: a new tool to diagnose infectious diseases
Abstract
This article presents a new use of the crispr-Cas genomic editing technology, which allowed Jennifer Doudna and Emmanuelle Charpentier to win the 2020 Nobel Prize in Chemistry. This tool not only allows the modification of dna sequences but can also be used as a very rapid and sensitive test for clinical diagnosis. Due to the recent pandemic, there has been an exponential growth in methods to detect sars-CoV-2, among which crispr-Cas stands out. The use of this technology allowed us to create a rapid and inexpensive test that can be easily implemented to increase the number of tests performed and thus contribute to a better control of the pandemic. Its role in molecular diagnosis also has great potential not only for detecting the presence of viruses and bacteria but also to diagnose non-infectious diseases such as cancer.
Keywords: genetic edition, diagnosis, detection, covid-19, sars-CoV-2.
Premio Nobel

Figura 1. Dra. Emmanuelle Charpentier (izquierda) y Dra. Jennifer Doudna (derecha). Modificada de: Bianca Fioretti of Hallbauer, Duncan Hull y The Royal Society.
El pasado 8 de diciembre de 2020, la Dra. Jennifer Doudna tuvo una de las más importantes ceremonias de premiación en el lugar menos esperado: el patio de su casa.
Ese día, la destacada bioquímica, en compañía de sólo 11 personas, recibió el premio Nobel en Química 2020 por sus contribuciones en el desarrollo de un método para la edición genómica denominado crispr-Cas (Clustered Regularly Interspaced Short Palindromic Repeats-Cas) (Jinek et al., 2012).
La Dra. Emmanuelle Charpentier, co-ganadora de este premio recibió de manera muy similar el galardón en la embajada de Suecia en Berlín (NobelPrize.org, 2021; ver figura 1).
La ceremonia, a la que asisten representantes de la prensa, televisión y un grupo grande de invitados, tuvo que ser escueta y en línea por la pandemia covid-19. Esto no le quita un ápice a la importancia del premio de este año.
La herramienta crispr-Cas ha revolucionado no sólo la investigación encaminada a descubrir los importantes mecanismos básicos de la vida, sino que también ha abierto la posibilidad de modificar el genoma1 humano con fines diagnósticos y terapéuticos. Esta última posibilidad ha generado muchas expectativas y controversias, debido a cuestiones éticas fundamentales, como la posibilidad de modificar el genoma humano de manera heredable.
¿Qué es CRISPR-Cas?
crispr-Cas, también llamado cotidianamente como “tijeras genéticas”, es un método de defensa que tienen algunas bacterias para protegerse de los virus. Los virus han coexistido con las bacterias desde el principio de su evolución, por lo que no es de extrañar que en estas últimas exista un mecanismo para defenderse. Dada la coevolución de virus y bacterias, hay muchas variaciones de estos sistemas de defensa. crispr-Cas es, entonces, el sistema inmune de las bacterias. Debido a que se trata de organismos unicelulares, no pueden tener células especializadas para producir anticuerpos, por lo que la manera en que logran defenderse es creando un sistema que reconoce al invasor, copia una parte de su material genético y lo guarda en su propio adn.2 Si vuelve a haber contacto con el mismo virus, la bacteria lo reconoce gracias a esa información almacenada, y lo inactiva al cortar su material genético. Es decir, funciona como el antivirus de una computadora, pues busca un código de programación específico (adn viral) y copia una parte a su disco duro (adn), para poder reconocerlo si vuelva a aparecer, en cuyo caso lo destruye (crispr-Cas).
¿Cómo funciona?
Luego del primer contacto, la bacteria selecciona un fragmento de adn que le permita identificar al virus. El método que utiliza para guardar la información de los distintos virus implica la inserción del fragmento viral en su propio adn, en una región especial para ello. En esta región, y con el fin de mantener un “archivo” de las secuencias de los virus, la bacteria usa separadores entre ellas, que son secuencias cortas en palíndromo de adn,3 que se presentan en intervalos regulares. De ahí vienen las siglas de crispr (Clustered Regularly Interspaced Short Palindromic Repeats). Posteriormente, ante otra infección con el virus, la bacteria producirá una molécula de arn4 a partir de esta secuencia en palíndromo.
Otro componente importante del sistema crispr es la enzima5 denominada Cas (Crispr ASsociated protein). Esta enzima forma un complejo con el arn producido por el palíndromo, que ya previamente incorporó la bacteria, y con el fragmento de genoma del virus infectante, al que llamamos guía (ambas secuencias se reconocen ya que son complementarias). Al encajar perfectamente el arn guía con el adn del virus, la enzima Cas activa su función de nucleasa,6 que es el nombre que se le da a la actividad de realizar cortes en los ácidos nucleicos (adn o arn), y corta el adn viral en fragmentos, destruyéndolo y por ende evitando la infección en la bacteria (ver figura 2).
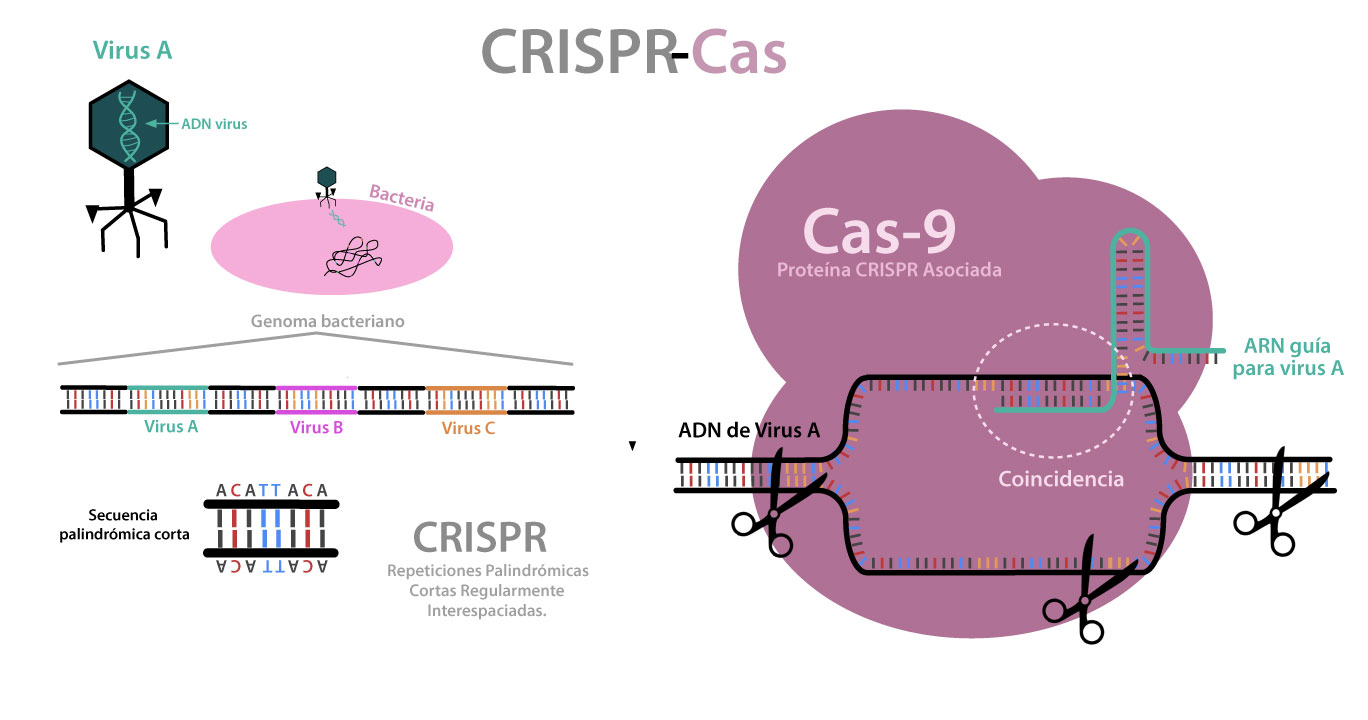
Figura 2. Sistema CRISPR-Cas9.
Una vista global
Un panorama de infinitas posibilidades se abrió ante la comunidad científica con el descubrimiento de este sistema, ya que permite reconocer cualquier secuencia de adn y cortar en ese sitio particular, para insertar una secuencia nueva en el corte o hacer mutaciones muy específicas.
Pensemos en una mutación que genera alguna enfermedad rara: con este procedimiento, que además resulta económico y muy eficiente, podríamos reparar el gen defectuoso. Este avance se puede realizar en el cuerpo adulto, pero también en embriones humanos. Esto último tiene fuertes implicaciones bioéticas, ya que se desconocen los posibles efectos colaterales y a largo plazo, así como la posibilidad de crear una modificación que afectará a todos los descendientes de la persona. Hasta ahora existen trabajos que permiten crear mutantes en algunos animales de laboratorio para ser usados como modelos de estudio de algunas enfermedades, así como trabajos iniciales en humanos, en donde se está estudiando la posibilidad de curar un tipo de ceguera.
Uso alternativo
Algo que ha recibido menos atención es el uso de la herramienta crispr-Cas para realizar diagnósticos moleculares con una sensibilidad enorme. Este sistema es tan flexible que nos permite utilizar diversas enzimas o modificaciones de éstas, abriendo un abanico gigantesco de posibles aplicaciones.
En este sentido, existe una familia de sistemas crispr-Cas derivadas de diferentes bacterias, que se dividen principalmente en dos clases. En la clase 1 encontramos a la Cas 6 y en la clase 2 encontramos a la Cas9, Cas12 y Cas13, que pueden reconocer adn (de doble cadena) y arn (de cadena sencilla), respectivamente (Koonin y Makarova, 2019; ver figura 3). Estas últimas son las más utilizadas como herramientas de edición. Las diferencias entre sus mecanismos han revolucionado el campo del diagnóstico molecular, dado a que es posible utilizarlas para detectar no sólo virus y bacterias, sino también enfermedades no infecciosas como el cáncer.
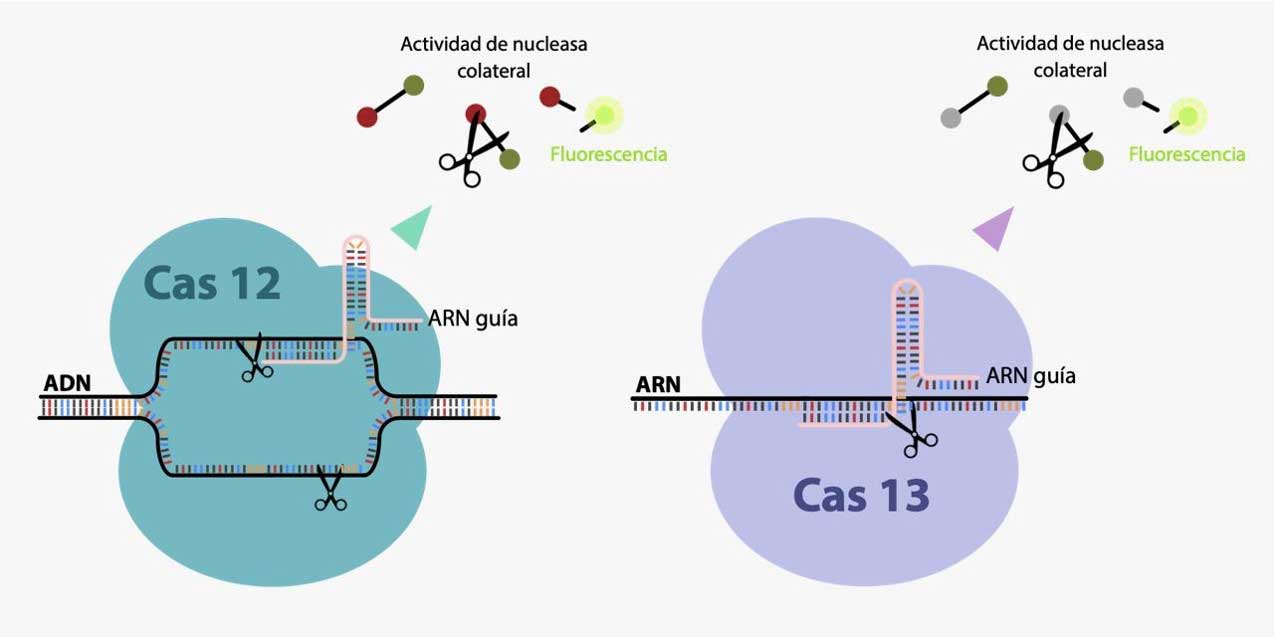
Figura 3. Sistema CRISPR-Cas.
El principio que se utiliza para el diagnóstico es básicamente el mismo: ambas enzimas cortan el fragmento deseado de adn o arn específico, luego de ser reconocido por el arn guía. La principal diferencia con Cas9 es que estas enzimas, al ser activadas, cortan de manera no específica todo el material genético que se encuentre cercano. Si al arn guía lo marcamos con una molécula “reportera”, que tenga fluorescencia, al ser cortada se liberará ésta, que emitirá una señal que podrá detectarse de diferentes maneras (ver figura 2). De esta manera, podemos detectar indirectamente secuencias de adn específicas.
Esta tecnología puede, entonces, utilizarse para detectar diminutas cantidades de adn en fluidos corporales o incluso en aguas residuales (Zhang, Kitajima, Whittle y Liu, 2017). Ejemplo de esto es la detección de fragmentos de adn mutado, provenientes de pacientes con cáncer, la detección rápida de organismos en el campo y de patógenos humanos, como malaria o zika. Un caso actual de mucho interés es su uso para detectar el virus de la covid-19 en aguas residuales de diversas ciudades, con el fin de determinar los niveles de la enfermedad y hacer un seguimiento en tiempo real.
Diagnóstico de SARS-CoV-2
Es curioso que la ceremonia de entrega del premio Nobel del 2020 haya tenido que ser en línea debido a la pandemia, porque justamente el desarrollo de la herramienta que les permitió a las doctoras Doudna y Charpentier obtener este premio puede ser utilizada para ayudar al mundo a salir de la emergencia sanitaria al usarla como herramienta de detección.
El virus causante de la covid-19, el sars-CoV-2, es un patógeno que está compuesto por arn, proteínas y una cápsula de la que emergen otras proteínas llamadas espiga por su forma afilada (spike en inglés), las cuales se encuentran en la superficie y le confieren al virus la apariencia de corona. Asimismo, estas mismas espigas le permiten infectar células humanas (Hu, Guo, Zhou y Shi, 2021) y son muy importantes en las vacunas contra este virus.
Durante la presente pandemia, se ha demostrado la utilidad del diagnóstico molecular de virus sars-CoV-2 mediante la prueba de rt-pcr7 cuantitativa (ver figura 4). Esta prueba ha permitido no sólo diagnosticar a los pacientes contagiados, sino llevar un control del número de infecciones y su distribución, para poder llevar a cabo las medidas de salud pública requeridas. Sin embargo, esta prueba tiene dos problemas principales: el costo y el tiempo requerido para realizarla. Con el fin de buscar alternativas, varios grupos de investigación han probado otras estrategias para solventar este problema, lo que ha llevado al uso de la técnica de crispr-Cas para detectar el virus de sars-CoV-2.
En nuestro grupo de investigación, buscamos paliar el problema del costo y tiempo de ejecución de la prueba de pcr mediante el uso de crispr-Cas (Garcia-Venzor et al., 2021). En primera instancia, nuestra intención fue eliminar la necesidad de extraer el arn del virus del hisopado (muestras tomadas de la garganta y parte posterior de la nariz) antes de realizar la prueba, dado a que el mayor costo y tiempo se generan durante este paso en la prueba convencional de rt-pcr. Tratamos varias aproximaciones para finalmente lograr detectar el arn del virus directamente de la muestra sin necesidad de hacer un paso de extracción del rna.
La prueba diseñada por nuestro grupo parte de una pequeña muestra nasal, a partir de la cual se libera el arn viral mediante calentamiento de apenas 25 minutos, lo que logra evitar el sistema de extracción usado en las pruebas actuales. Después, el arn liberado se convierte a una hebra de adn, para que sea más estable, y posteriormente se amplifica o multiplica una secuencia específica del virus, con el fin de obtener una cantidad suficiente para poder ser detectada después. Esta amplificación no utiliza la tecnología de la pcr, sino que se realiza mediante un sistema conocido como lamp,8 que es una técnica sumamente rápida en comparación con una pcr y con mayor eficiencia. Utiliza una única temperatura durante todo el proceso, por lo que no es necesario tener equipos sofisticados para realizarla, puede llevarse a cabo incluso en un baño maría,9 que mantenga la temperatura fija, lo cual resulta accesible pues se trata de materiales de cabecera en muchos laboratorios.
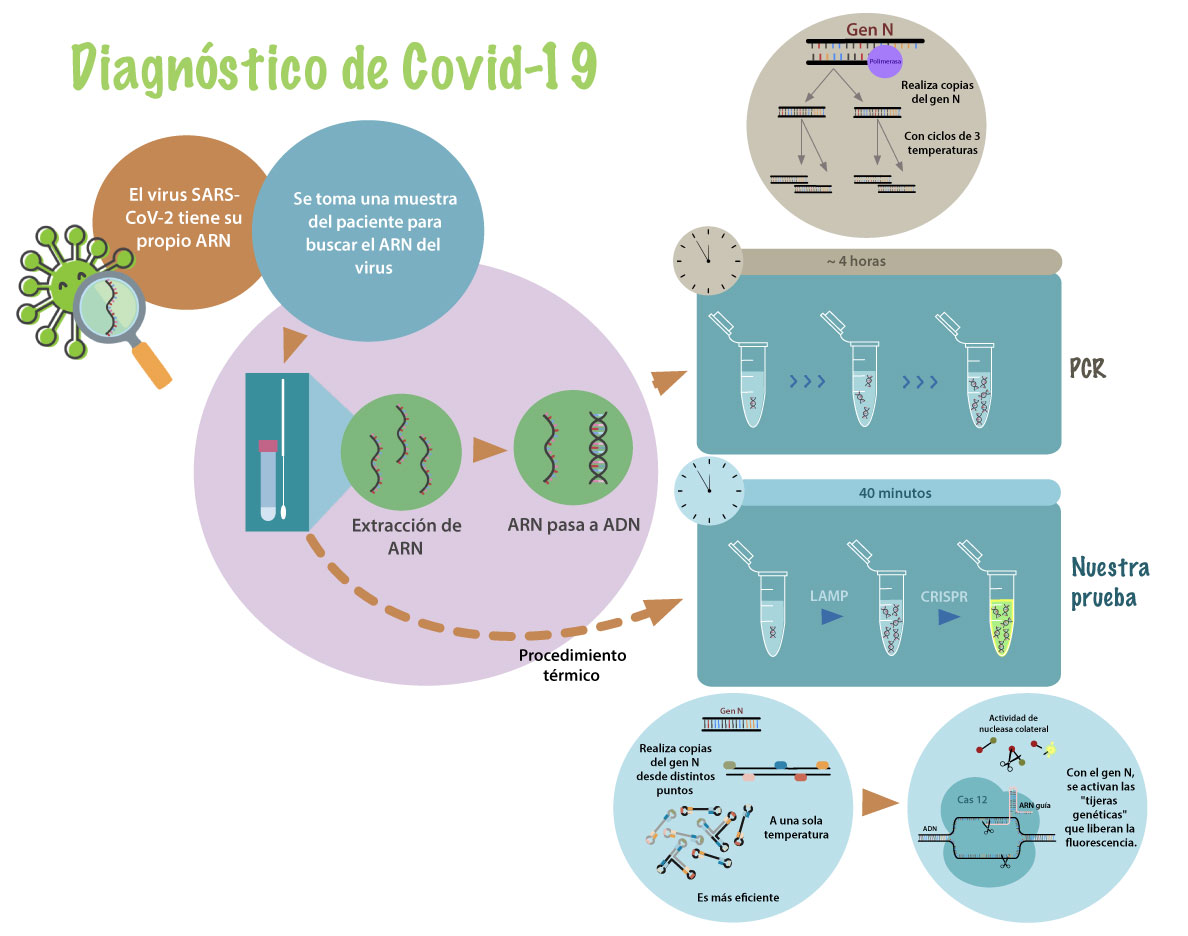
Figura 4. Sistema rt-pcr para detectar SARS-CoV-2.
Finalmente, el adn amplificado muchas veces se utiliza para detectar la presencia del virus mediante un sistema crispr/Cas12. En éste tenemos la enzima Cas12 previamente unida al arn guía, el cual tiene como blanco un gen particular del virus: el gen N. Este gen se ha utilizado en ambas pruebas, ya que su secuencia es específica de sars-CoV-2 y no se cruza ni con otro virus de la misma familia. Una vez que el complejo de Cas12-arn guía reconoce su secuencia blanco, se activa el dominio de nucleasa colateral. Este dominio comienza a cortar indistintamente moléculas de adn. El reportero mencionado anteriormente es un fragmento de adn, que contiene en un extremo una molécula fluorescente (llamada fluorocromo) y en el otro una molécula que lo neutraliza al absorber la luz emitida por el fluorocromo. Al cortarse este fragmento, el fluorocromo queda libre para emitir fluorescencia. Esto nos permite detectar la presencia del virus mediante la emisión de luz fluorescente, es decir con la emisión de un brillo verdoso que puede ser detectado a simple vista al incidir una luz azul sobre la muestra (ver figura 3).
Comentario final
crispr-Cas es una herramienta que se sigue explorando con la intención de buscar aplicaciones novedosas para resolver distintos problemas a nivel genómico. El uso de este sistema como método diagnóstico nos permite tener pruebas rápidas y económicas que pueden ayudarnos no sólo a mejorar el diagnóstico de enfermedades emergentes, sino a realizar un monitoreo epidemiológico más preciso y en tiempo real. En un futuro esperamos que sea posible realizar estas pruebas para las enfermedades infecciosas endémicas que aquejan a nuestra población, permitiendo destinar recursos económicos y materiales de manera focalizada y directa.
Referencias
- Garcia-Venzor A., Rueda-Zarazua, B., Marquez-Garcia, E., Maldonado, V., Moncada-Morales, A., Olivera, H., Lopez, I., Zuñiga, J. y Melendez-Zajgla, J. (2021). sars-CoV-2 Direct Detection Without rna Isolation With Loop-Mediated Isothermal Amplification (lamp) and crispr-Cas12. Frontiers in Medicine, 8(125). https://doi.org/10.3389/fmed.2021.627679.
- Jinek, M., Chylinskiines, K., Fonfara, I., Hauer, M., Doudna, J. A. y Charpentier, E. (2012). A programmable dual-rna-guided dna endonuclease in adaptive bacterial immunity. Science, 337(6096), 816-821. https://doi.org/10.1126/science.1225829.
- NobelPrize. (2021). The Nobel Prize in Chemistry 2020. https://www.nobelprize.org/prizes/chemistry/2020/summary/.
Recepción: 25/02/2021. Aprobación: 21/04/2021.


