Armas moleculares bacterianas: el sistema de secreción tipo 3
Vol. 23, núm. 4 julio-agosto 2022
Armas moleculares bacterianas: el sistema de secreción tipo 3
Luis Fernando Montelongo Martínez y Miguel Cocotl Yañez CitaResumen
La mayoría de las bacterias son benéficas para los humanos, sin embargo, algunas pueden causarnos enfermedades, ya que producen una serie de compuestos, conocidos como factores de virulencia, que nos dañan. Uno de los principales determinantes de virulencia es el sistema de secreción tipo iii (sst3), que las bacterias usan como un arma molecular que les permite inyectar toxinas a nuestras células y, con ello, llevarlas a la muerte. Una de estas bacterias es Pseudomonas aeruginosa, un patógeno oportunista, que puede ser resistente a diversos antibióticos. Además, esta bacteria se coordina y comunica mediante los sistemas de detección de quorum (qs) que le permiten, entre otras cosas, activar su virulencia cuando son suficientes en número. Un determinante crítico de su virulencia es el sst3 cuya activación depende de la proteína ExsA, la cual es regulada a nivel transcripcional y traduccional por los sistemas de qs y Rsm, respectivamente. El sst3 es tan importante en esta bacteria que si no se activa disminuye su virulencia; por eso es importante entender los mecanismos que controlan su expresión, con el fin de diseñar estrategias para evitar su activación y, con ello, ayudar al tratamiento de las infecciones causadas por este microorganismo.
Palabras clave: Pseudomonas, virulencia, bacterias, sensor de quórum, RsmA, sistema de secreción tipo 3.
Molecular weapons: the type III secretion system
Abstract
Most bacteria are beneficial to humans but some of them are able to infect us and cause disease, due to virulence factors. An important virulence factor is the type iii secretion system (t3ss) that bacteria use as a molecular weapon that allows them to inject toxins into our cells causing death. One of these bacteria is Pseudomonas aeruginosa, an opportunistic pathogen that can be resistant to multiple antibiotics. Moreover, these bacteria can communicate with each other and coordinate a group behavior by the quorum sensing systems (qs) that activates the production of virulence factors. The t3ss activation depends upon on the ExsA protein that is regulated at transcriptional and translational levels by the qs and Rsm systems, respectively. Since inactivation of the t3ss impairs the virulence of this bacterium, it is important to understand the mechanisms that control its expression, which will allow the design of strategies to avoid its activation and thus assist to the treatment of the infections caused by this microorganism.
Keywords: Pseudomonas, virulence, bacteria, quorum sensing, RsmA, type III secretion system.
Introducción
Comencemos por mencionar que la mayoría de las bacterias que habitan en el ambiente no son patógenas para el ser humano, de hecho, muchas de ellas son benéficas para nosotros, y otras tantas son necesarias para que podamos vivir (Marchesi et al., 2016). Sin embargo, dentro de esa diversidad bacteriana, se encuentran aquellas que son capaces de infectarnos y provocarnos enfermedades, pero para lograrlo deben sobrepasar todas las barreras que tenemos, como el sistema inmune. Por lo tanto, a pesar de ser organismos microscópicos con un tamaño promedio de 5 micrómetros (es decir 0.000005 metros), las bacterias son capaces de coordinarse para ocasionar una infección. No sólo eso, las bacterias también poseen armas moleculares que les permiten competir con otras bacterias por el hábitat y nutrientes o incluso combatir a nuestro sistema inmune cuando ese nicho somos nosotros.
Dentro de las armas moleculares que poseen estos entes microscópicos se encuentran unas estructuras complejas, ancladas a sus membranas celulares, que les permiten expulsar, de manera controlada, componentes con una amplia variedad de funciones, llamados sistemas de secreción (ss; Green y Mecsas, 2016). Dentro de las moléculas secretadas se encuentran las exotoxinas, moléculas que son dañinas para nuestras células, y que son producidas por algunas bacterias patógenas, por lo cual son determinantes en la virulencia del microorganismo. Por lo tanto, debido a la importancia de los ss, las exotoxinas se han convertido en blancos terapéuticos en el tratamiento de infecciones.
El sistema de secreción tipo 3
Hasta la fecha, se han identificado ocho tipos de ss, algunos de los cuales están conservados en los tres dominios de la vida (Archea, Bacteria y Eucaria, basados en las diferencias en la secuencia del arn ribosomal). Estos tipos de ss han sido definidos en su estructura composición y actividad. Generalmente están inactivos, pero responden a cambios en las condiciones del medio donde se encuentran las bacterias. Su activación cumple el reto bioquímico de secretar componentes, como las exotoxinas, a través de las membranas impermeables. Estas exotoxinas poseen funciones específicas, que favorecen la adaptación, competencia y virulencia bacterianas (Rüter et al., 2018; Dey et al., 2019).
Uno de los sistemas que representa mayor interés en la investigación es el sistema de secreción tipo 3 (sst3), también llamado inyectisoma. Curiosamente, su estructura y función son similares a las de una jeringa convencional, como las usadas por los médicos para inyectarnos. Estas máquinas nanomoleculares son utilizadas por una amplia variedad de bacterias patógenas y simbiontes (aquellos que establecen relaciones de beneficio mutuo) de plantas, mamíferos, hongos y protozoarios. Sin duda, son uno de los factores de virulencia más sofisticados en la naturaleza; aunque su origen es aún tema de controversia, se sabe que tienen relación evolutiva con el flagelo bacteriano (la estructura bacteriana que les permite desplazarse). La hipótesis es que, a lo largo de la evolución, el flagelo cambió su función de rotar para brindarle movilidad a la bacteria, por una función secretora que apoya su establecimiento y colonización del territorio (Deng et al., 2017).
Los sst3 son tan importantes en la virulencia bacteriana que han sido pieza fundamental de las grandes epidemias en la historia de la humanidad. Un ejemplo es la peste negra, la cual diezmó a más de un tercio de la población de toda Europa en la Edad Media. Esta plaga fue causada por la bacteria Yersinia pestis, la cual se transmite al ser humano por la mordedura de pulgas de roedores infectados. Su principal arma molecular es el sst3, que le permite inyectar toxinas a nuestras células, o que potencialmente puede desencadenar su muerte (Plano y Schesser, 2013). Además de Y. pestis, otras bacterias patógenas para el hombre, que también usan el sst3 como arma molecular, incluyen a Salmonella enterica, Shigella flexneri, Escherichia coli (causantes de infecciones gastrointestinales) y Pseudomonas aeruginosa (asociada a infecciones del tracto urinario, oculares, del oído, entre otras; ver figura 1).
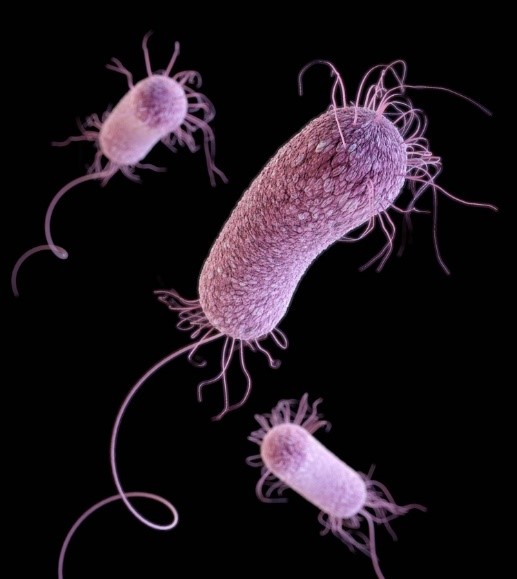
Figura 1. Imagen tridimensional, por computadora, de Pseudomonas aeruginosa, multirresistentes a antibióticos, basada en imágenes generadas por microscopio electrónico de barrido.
Crédito: Oosthuizen, 2013.
Pseudomonas aeruginosa y el SST3
P. aeruginosa es una bacteria versátil que se encuentra en varios ambientes, ya que puede crecer utilizando diferentes fuentes de carbono. Su importancia radica en que es una bacteria patógena para el hombre, pues infecta y ocasiona enfermedad en pacientes inmunocomprometidos, que han sufrido quemaduras graves o pacientes que sufren de fibrosis quística, una enfermedad hereditaria que provoca una acumulación excesiva de moco en los pulmones y que llega a ser mortal. No sólo eso, esta bacteria es naturalmente resistente a diversos antibióticos y es capaz de adquirir nuevas resistencias, lo que genera un problema, debido a que el tratamiento para eliminarla puede complicarse (Azam y Khan, 2019). A pesar de que esta bacteria posee un amplio repertorio de armas moleculares, destaca entre ellas el sst3.
El sst3 de P. aeruginosa es un complejo de más de 25 proteínas, las cuales son codificadas por 36 genes ampliamente conservados en diferentes bacterias (Hauser, 2009). Este inyectisoma le permite a la bacteria introducir toxinas mediante la formación de un canal que atraviesa membranas celulares bacterianas, pared celular y que finalmente perfora la membrana celular del hospedero (sí, nuestras células), donde son liberadas las toxinas.
Los principales componentes del sst3 son un aparato exportador, que reconoce a las toxinas; el cuerpo basal, que se compone de proteínas que lo anclan a las membranas; un filamento (que funciona como una aguja), y un componente translocador, que se une a la membrana de las células diana para formar un poro, en el cual se acopla la aguja y de esta manera se crea un canal por donde se transportan las toxinas desde el citoplasma bacteriano al citoplasma hospedero, proceso muy similar a una inyección en nuestro brazo (ver figura 2). Aunque la estructura del sst3 está conservada, la cantidad y función de las toxinas son exclusivas de cada bacteria. Existen bacterias patógenas que son capaces de sintetizar y secretar decenas de efectores con diferentes funciones. No obstante, la capacidad de sintetizar varias toxinas no garantiza el poderío infeccioso. Ejemplo de ello es P. aeruginosa, de la cual se han identificado sólo cuatro exotoxinas llamadas ExoS, ExoU, ExoT y ExoY que son suficientes para la virulencia de la bacteria.
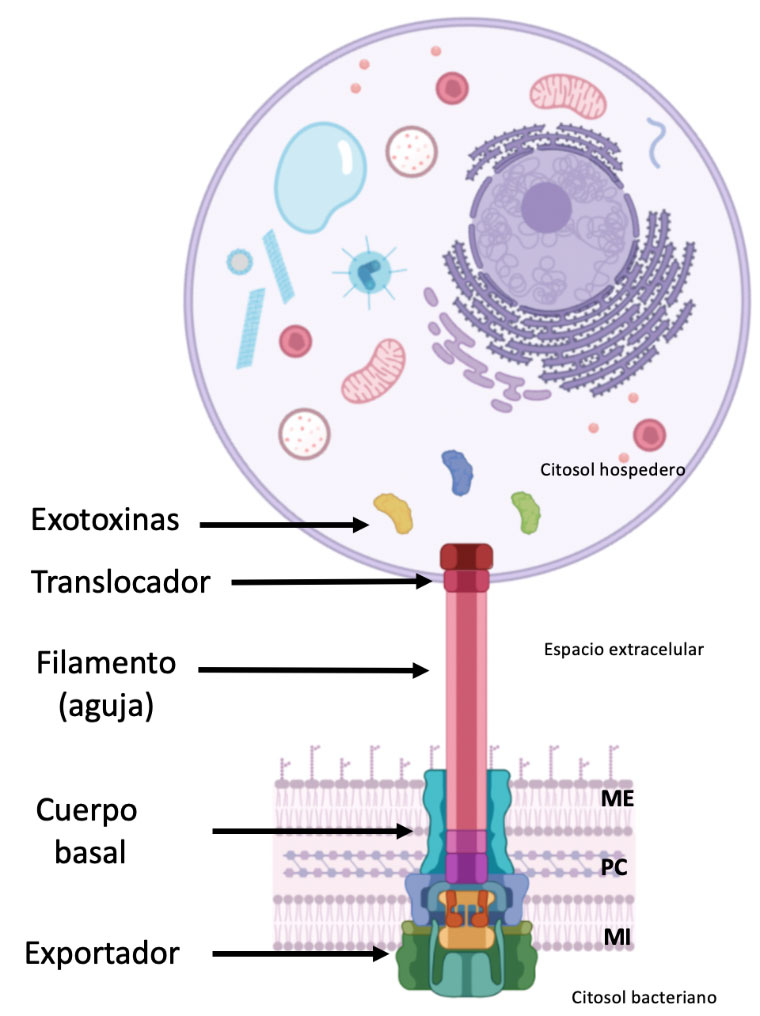
Figura 2. Estructura del SST3 de P. aeruginosa. Sus principales componentes son: exportador, cuerpo basal, filamento, translocador y exotoxinas. ME es la membrana externa, PC es la pared celular y MI, la membrana interna.
Figura creada en Biorender.com.
Las toxinas son secretadas en su forma inactiva, lo que evita que sean dañinas para la bacteria que los sintetiza. Así, éstas sólo se activan al entrar en contacto con componentes citoplasmáticos de la célula que infectan. Las toxinas activas tienen la capacidad de interferir cascadas de señalización intracelulares, modificar el citoesqueleto (red de proteínas que dan soporte y forma a la célula), inhibir la fagocitosis (proceso que permite a diversas células del sistema inmunológico atrapar y eliminar patógenos), e inducir la muerte celular programada y la necrosis celular (procesos en los que nuestras células mueren).
El éxito del ataque se basa en la capacidad de la bacteria para controlar la síntesis y secreción de los componentes del sst3. Para ello, la bacteria posee una proteína reguladora, llamada ExsA, que activa todos los genes necesarios para el ensamblaje y función del sst3. La inactivación del gen exsA evita la formación y actividad del sst3, por lo cual la virulencia de la bacteria disminuye. Por lo tanto, debido a la importancia de exsA, su expresión se encuentra finamente controlada a nivel de su transcripción y traducción (ver figura 3).
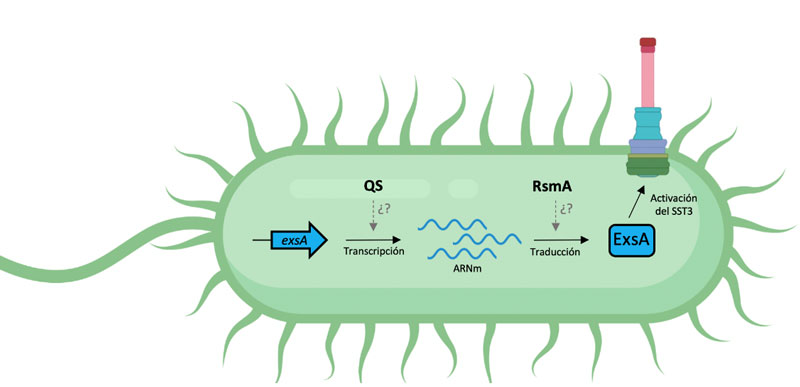
Figura 3. ExsA activa al SST3. El inyectisoma es regulado positivamente por ExsA cuya expresión es regulada a nivel transcripcional por los sistemas de detección de quorum (QS) y a nivel traduccional por RsmA.
Figura creada en Biorender.com.
Respecto a la regulación a nivel transcripcional, es decir la conversión de adn a arn mensajero (arnm), ésta involucra a los sistemas de detección de quórum (qs por sus siglas en inglés) que posee P. aeruginosa. Estos sistemas son un tipo de lenguaje bacteriano que le permite a Pseudomonas detectar a otras Pseudomonas que están a su alrededor, mediante la producción de moléculas señal conocidas como autoinductores (ai), que son secretadas al medio. Cuando hay un gran número de bacterias, y por lo tanto una acumulación de ai, estas moléculas señal entran nuevamente a la célula, lo que permite la activación de proteínas reguladoras capaces de encender o apagar la expresión de genes de manera directa o indirecta. P. aeruginosa posee tres tipos de sistemas de qs llamados Las, Rhl y Pqs, organizados de manera jerárquica, según las condiciones de cultivo. Estos sistemas de qs regulan hasta 10% del total de los genes de la bacteria, principalmente los de virulencia, incluyendo genes del sst3 (Hogardt et al., 2004).
Respecto a la regulación a nivel de la traducción, es decir el paso del arnm a proteína, ésta involucra a los sistemas Gac-Rsm. Gac es un sistema de doble componente, que detecta señales fuera de la célula para controlar la expresión de unos arn pequeños conocidos como RsmV, RsmW, RsmY y RsmZ, que no codifican para proteínas y los cuales secuestran a una proteína llamada RsmA, que es capaz de unirse a arnm y regular su traducción. En el caso del sst3, RsmA regula de manera positiva la expresión de exsA (Brencic y Lory, 2009). Así, cuando la señal que el sistema Gac detecta no se encuentra presente en el medio (y que aún es desconocida), entonces, no se enciende la expresión de los arn pequeños y RsmA se encuentra libre para activar al sst3 y, por lo tanto, la secreción de las exotoxinas a las células blanco.
Así, se ha propuesto que los sistemas Gac y Rsm actúan como un interruptor que controla el paso de una infección aguda (que involucra la activación del sst3) a una crónica.1 Además de estos sistemas globales de regulación, existen otros reguladores que también están controlando la expresión del sst3 lo que indica su importancia (McMackin et al., 2019). En nuestro grupo de investigación estamos elucidando los mecanismos por los cuales los sistemas de qs y el sistema Rsm controla la expresión de los genes de este sistema de secreción en P. aeruginosa.
Finalmente, aunque el sst3 ha sido estudiado ampliamente en P. aeruginosa, aún quedan por responder varias preguntas, como el hecho de conocer en detalle las señales que activan al sst3 dentro del cuerpo humano. Hasta ahora sólo se ha determinado que la baja concentración de calcio es uno de los requerimientos para que el sistema se active. Se desconoce cómo la bacteria identifica en qué momento iniciar la inyección de las exotoxinas, o por qué tiene cierta preferencia por inyectar las exotoxinas a ciertos tipos celulares, como las células del sistema inmune. Al conocer con mayor detalle todos estos mecanismos, será más fácil plantear estrategias para evitar su activación y, por lo tanto, se podrá reducir la virulencia de esta bacteria o incluso de otras que tienen mecanismos similares. Esto también permitirá coadyuvar al tratamiento de las infecciones causadas por este microorganismo.
Referencias
- Azam, M. W., y Khan, A. U. (2019). Updates on the pathogenicity status of Pseudomonas aeruginosa. Drug Discovery Today, 24(1), 350-359. https://doi.org/10.1016/j.drudis.2018.07.003.
- Brencic, A., y Lory, S. (2009). Determination of the regulon and identification of novel mRNA targets of Pseudomonas aeruginosa RsmA. Molecular Microbiology, 72(13), 616-632. https://doi.org/10.1111/j.1365-2958.2009.06670.x.
- Deng, W., Marshall, N. C., Rowland, J. L., McCoy, J. M., Worrall, L. J., Santos, A. S., Strynadka, N. C. J., y Finlay, B. B. (2017). Assembly, structure, function, and regulation of type iii secretion systems. Nature Review Microbiology, 15(6), 323-337. https://doi.org/10.1038/nrmicro.2017.20.
- Dey, S., Chakravarty, A., Guha Biswas, P., y De Guzman, R. N. (2019). The type iii secretion system needle, tip, and translocon. Protein Science, 28(9),1582-1593. https://doi.org/10.1002/pro.3682.
- Green, E. R., y Mecsas, J. (2016). Bacterial Secretion Systems: An Overview. Microbiology Spectrum, 4(1), https://doi.org/gf439f.
- Hauser, A. R. (2009). The type iii secretion system of Pseudomonas aeruginosa: infection by injection. Nature Review Microbiology, 7(9), 654-665. https://doi.org/10.1038/nrmicro2199.
- Hogardt, M., Roeder, M., Schreff, A. M., Eberl, L., y Heesemann, J. (2004). Expression of Pseudomonas aeruginosa exoS is controlled by quorum sensing and RpoS. Microbiology (Reading), 150(Pt 4), 843-851. https://doi.org/10.1099/mic.0.26703-0.
- Marchesi, J. R., Adams, D. H., Fava, F., Hermes, G. D. A., Hirschfield, G. M., Hold, G., Quraishi, M. N., Kinross, J., Smidt, H., Tuohy, K. M., Thomas, L. V., Zoetendal, E., y Hart, A. (2016). The gut microbiota and host health: a new clinical frontier. Gut, 65(2), 330-9. https://doi.org/10.1136/gutjnl-2015-309990.
- McMackin, E. A. W., Djapgne, L., Corley, J. M., y Yahr, T. L. (2019). Fitting Pieces into the Puzzle of Pseudomonas aeruginosa Type iii Secretion System Gene Expression. Journal Bacteriology, 201(13), e00209-19. https://doi.org/10.1128/JB.00209-19.
- Oosthuizen, J. (2013). Multidrug-resistant Pseudomonas aeruginosa. Public Health Image Library, Centers for Disease Control and Prevention. https://phil.cdc.gov/Details.aspx?pid=16876.
- Plano, G. V., y Schesser, K. (2013). The Yersinia pestis type iii secretion system: expression, assembly and role in the evasion of host defenses. Immunologic Research, 57(1-3), 237-45. https://doi.org/10.1007/s12026-013-8454-3.
- Rüter, C., Lubos, M. L., Norkowski, S., y Schmidt, M. A. (2019). All in-Multiple parallel strategies for intracellular delivery by bacterial pathogens. International Journal of Medical Microbiology, 308(7), 872-881. https://doi.org/10.1016/j.ijmm.2018.06.007.
Recepción: 05/08/2021. Aprobación: 18/05/2022.


